Preparativos
Carga de paquetes
Conjuntos de datos utilizados
Provincias de Costa Rica# Carga de la capa de provincias
provincias <-
st_read(
"https://raw.githubusercontent.com/gf0604-procesamientodatosgeograficos/2021i-datos/main/ign/delimitacion-territorial-administrativa/cr_provincias_simp_wgs84.geojson",
quiet = TRUE
)
# Mapa de la capa de provincias
plot(
provincias$geometry,
extent = extent(-86,-82.3, 8, 11.3),
main = "Provincias de Costa Rica",
axes = TRUE,
graticule = TRUE
)
# Carga de la capa de cantones
cantones <-
st_read(
"https://raw.githubusercontent.com/gf0604-procesamientodatosgeograficos/2021i-datos/main/ign/delimitacion-territorial-administrativa/cr_cantones_simp_wgs84.geojson",
quiet = TRUE
)
# Mapa de la capa de cantones
plot(
cantones$geometry,
extent = extent(-86,-82.3, 8, 11.3),
main = "Cantones de Costa Rica",
axes = TRUE,
graticule = TRUE
)
# Carga de la capa de ASP
asp <-
st_read(
"https://raw.githubusercontent.com/gf0604-procesamientodatosgeograficos/2021i-datos/main/sinac/asp/asp-wgs84.geojson",
quiet = TRUE
)
# Mapa de la capa de ASP
plot(
asp$geometry,
extent = extent(-86,-82.3, 8, 11.3),
main = "Áreas silvestres protegidas (ASP) de Costa Rica",
axes = TRUE,
graticule = TRUE
)

# Carga de la capa de aeródromos
aerodromos <-
st_read(
"https://raw.githubusercontent.com/gf0604-procesamientodatosgeograficos/2021i-datos/main/ign/aerodromos/aerodromos-wgs84.geojson",
quiet = TRUE
)
# Mapa de la capa de aeródromos
plot(
aerodromos$geometry,
extent = extent(-86,-82.3, 8, 11.3),
main = "Aeródromos de Costa Rica",
axes = TRUE,
graticule = TRUE
)
# Carga de la capa de vipéridos
viperidos <-
st_read(
"https://raw.githubusercontent.com/gf0604-procesamientodatosgeograficos/2021i-datos/main/gbif/viperidae-cr-registros.csv",
options = c(
"X_POSSIBLE_NAMES=decimalLongitude",
"Y_POSSIBLE_NAMES=decimalLatitude"
),
quiet = TRUE
)
# Asignación del sistema de coordenadas
st_crs(viperidos) = 4326
# Mapa de la capa de vipéridos
plot(
viperidos$geometry,
pch = 16,
extent = extent(-86,-82.3, 8, 11.3),
main = "Registros de Viperidae (tobobas) en Costa Rica",
col = "green",
axes = TRUE,
graticule = TRUE
)
Introducción
Esta lección brinda una visión general de las operaciones espaciales en datos vectoriales implementadas en el paquete sf y en datos raster implementadas en el paquete raster.
Datos vectoriales
Las operaciones espaciales en datos vectoriales incluyen:
- Creación de subconjuntos espaciales (spatial subsetting).
- Cruce de datos espaciales (spatial joining).
- Agregación espacial (spatial aggregation).
- Relaciones de distancia.
Manejo de datos espaciales con el paquete sf
Creación de subconjuntos espaciales
Es el proceso de selección de objetos espaciales con base en su relación con otros objetos espaciales. Estas relaciones se expresan como predicados espaciales, los cuales están implementados como métodos de sf.
La creación de subconjuntos espaciales es análoga a la creación de subconjuntos por datos de atributos. Puede realizarse a través de los operadores [ y $ del paquete base de R o por medio de la función filter() de dplyr.
En los dos ejemplos siguientes, se utiliza el método st_within() para filtrar los puntos contenidos en un polígono. Primero, se utilizan los operadores del paquete base.
# Selección de la provincia de Limón (por atributos)
limon <- provincias[provincias$provincia == "Limón", ]
# Selección de los aeródromos ubicados en Limón (espacial)
aerodromos_limon <- aerodromos[limon, , op = st_within]
# Mapa de aeródromos
plot(
limon$geometry,
main = "Aeródromos de Limón (1)",
axes = TRUE,
graticule = TRUE,
reset = FALSE
)
plot(aerodromos_limon$geometry, pch=16, add = TRUE)
El mismo resultado se obtiene con las funciones y operadores de Tidyverse.
# Selección de la provincia de Limón (por atributos)
limon <-
provincias %>%
filter(provincia == "Limón")
# Selección de los aeródromos ubicados en Limón (espacial)
aerodromos_limon <-
aerodromos %>%
filter(st_within(x = ., y = limon, sparse = FALSE))
# Mapa de aeródromos
plot(
limon$geometry,
main = "Aeródromos de Limón (2)",
axes = TRUE,
graticule = TRUE,
reset = FALSE
)
plot(aerodromos_limon$geometry, pch = 16, add = TRUE)
En el anterior llamado a filter(), la expresión x = . es equivalente a x = aerodromos. Para una explicación sobre el argumento sparse, por favor lea la sección 4.2.2. del libro “Geocomputation with R” de R. Lovelace et. al..
Además de st_within(), sf implementa predicados espaciales como, entre otros, st_contains(), st_intersects() y st_disjoint(), los cuales se ejemplifican en el siguiente bloque de código.
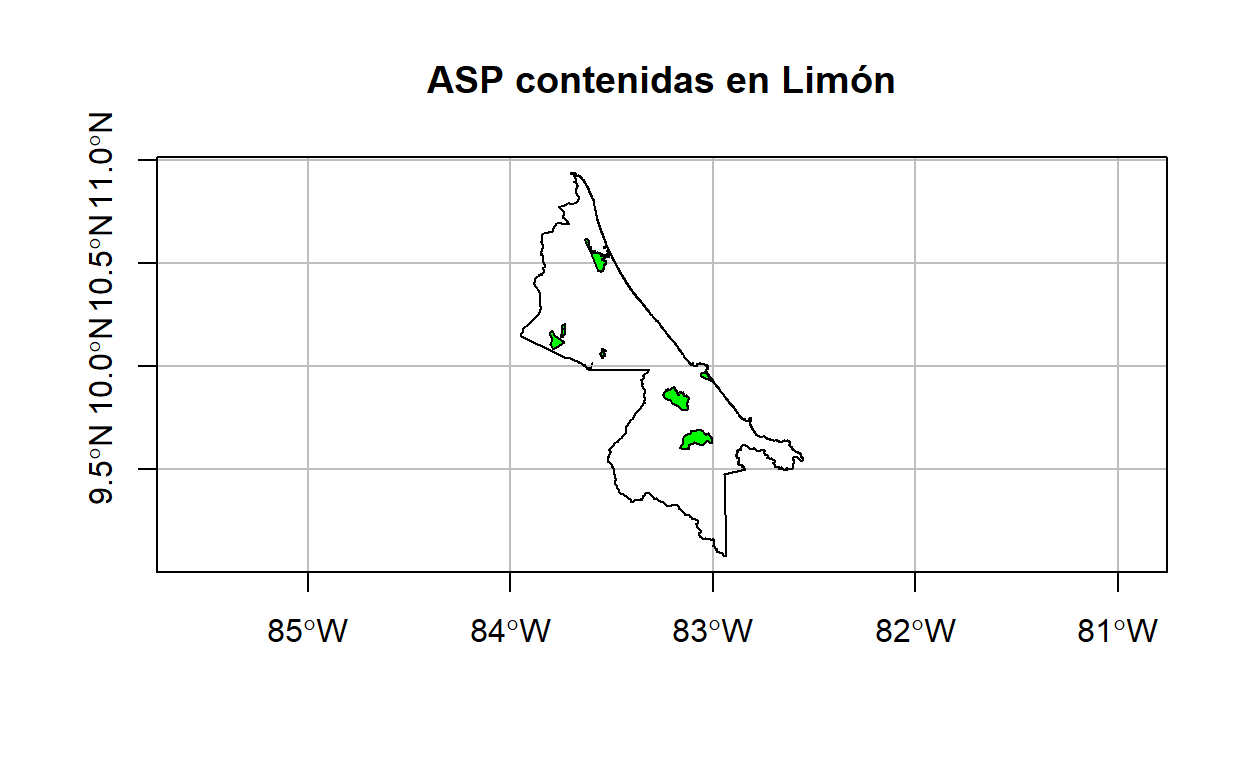
## Ejemplo de st_contains: ASP contenidas en Limón
# Selección de las ASP contenidas en Limón
asp_limon <-
asp %>%
filter(st_contains(x = limon, y = ., sparse = FALSE))
# Mapa de ASP contenidas en Limón
plot(
limon$geometry,
main = "ASP contenidas en Limón",
axes = TRUE,
graticule = TRUE,
reset = FALSE
)
plot(asp_limon$geometry, col = "green", add = TRUE)
## Ejemplo de st_intersects: ASP intersecadas con Limón
# Selección de las ASP intersecadas con Limón
asp_limon <-
asp %>%
filter(st_intersects(x = limon, y = ., sparse = FALSE))
# Mapa de ASP intersecadas con Limón
plot(
limon$geometry,
main = "ASP intersecadas con Limón",
axes = TRUE,
graticule = TRUE,
reset = FALSE
)
plot(asp_limon$geometry, col = "green", add = TRUE)

## Ejemplo de st_disjoint: ASP ubicadas fuera de Limón
# Selección de las ASP ubicadas fuera de Limón
asp_fuera_limon <-
asp %>%
filter(st_disjoint(x = limon, y = ., sparse = FALSE))
# Mapa de ASP ubicadas fuera de Limón
plot(
provincias$geometry,
extent = extent(-86,-82.3, 8, 11.3),
main = "ASP ubicadas fuera de Limón",
axes = TRUE,
graticule = TRUE,
reset = FALSE
)
plot(asp_fuera_limon$geometry, col = "green", add = TRUE)

Cruce de datos espaciales
El cruce “no espacial” de dos conjuntos de datos se basa en uno o varios campos (llamados llaves o keys) que están presentes en ambos conjuntos. Los cruces espaciales se basan en un principio similar pero, en lugar de campos comunes, la relación entre los conjuntos se realiza a través de una operación topológica, a veces llamada spatial overlay. Al igual que con los datos de atributos, el cruce espacial, ejecutado con el método st_join(), agrega una o varias columnas al conjunto de datos destino (i.e. el argumento x de la función), provenientes del objeto fuente (i.e. el argumento y).
En el siguiente ejemplo, se cruzan los registros de presencia de una especie (geometrías de puntos) con la capa de cantones (geomtrías de polígonos), para agregar la columna de nombre del cantón al conjunto de registros de presencia.
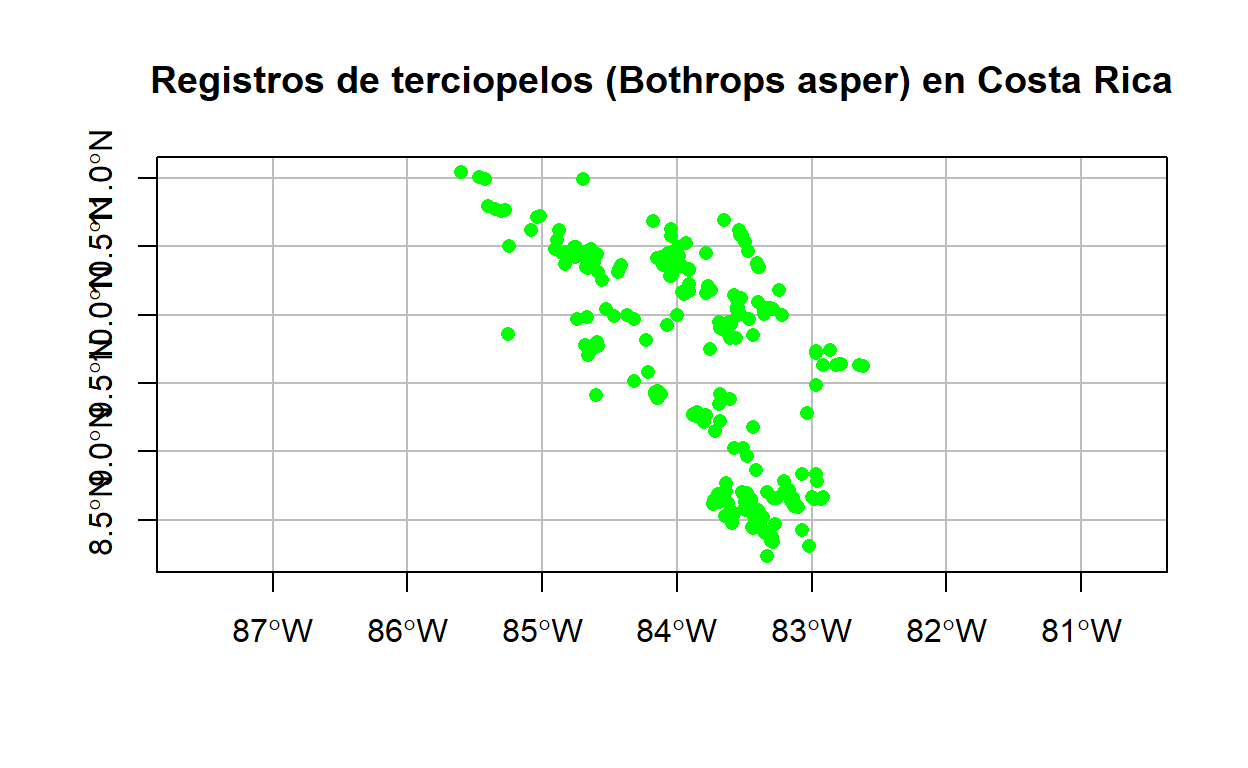
# Filtrado de los registros de serpientes de terciopelo (Bothrops asper) en el conjunto de vipéridos
terciopelos <-
viperidos %>%
filter(species == "Bothrops asper")
# Mapeo de la capa de terciopelos
plot(
terciopelos$geometry,
pch = 16,
main = "Registros de terciopelos (Bothrops asper) en Costa Rica",
col = "green",
axes = TRUE,
graticule = TRUE
)
# Cruce espacial con la tabla de cantones, para obtener el nombre del cantón
terciopelos <-
terciopelos %>%
st_join(cantones["canton"])
# Despliegue de los datos cruzados
terciopelos %>%
st_drop_geometry() %>%
slice(1:10) %>%
select(stateProvince, canton, locality)
stateProvince canton locality
1 Puntarenas <NA>
2 Limón Talamanca
3 Alajuela Upala
4 Limón Siquirres
5 Heredia Sarapiquí
6 Puntarenas Osa
7 Heredia Sarapiquí
8 Heredia Sarapiquí
9 Guanacaste Liberia
10 Alajuela Upala La función st_join() realiza por defecto un left join, pero puede realizar cruces de otros tipos también. Por ejemplo, con el argumento left = FALSE, puede realizarse un inner join. También por defecto, la operación topológica que se aplica es st_intersects().
Agregación de datos espaciales
De manera similar al caso de la agregación de atributos, la agregación espacial es una forma de “condensar” o “resumir” datos. Los datos agregados muestran estadísticas de una variable (ej. promedio, suma) en relación con una variable de agrupación. Esto puede lograrse con el método agregate() de sf o con los métodos group_by() y summarize() de dplyr, utilizados en combinación con st_join().
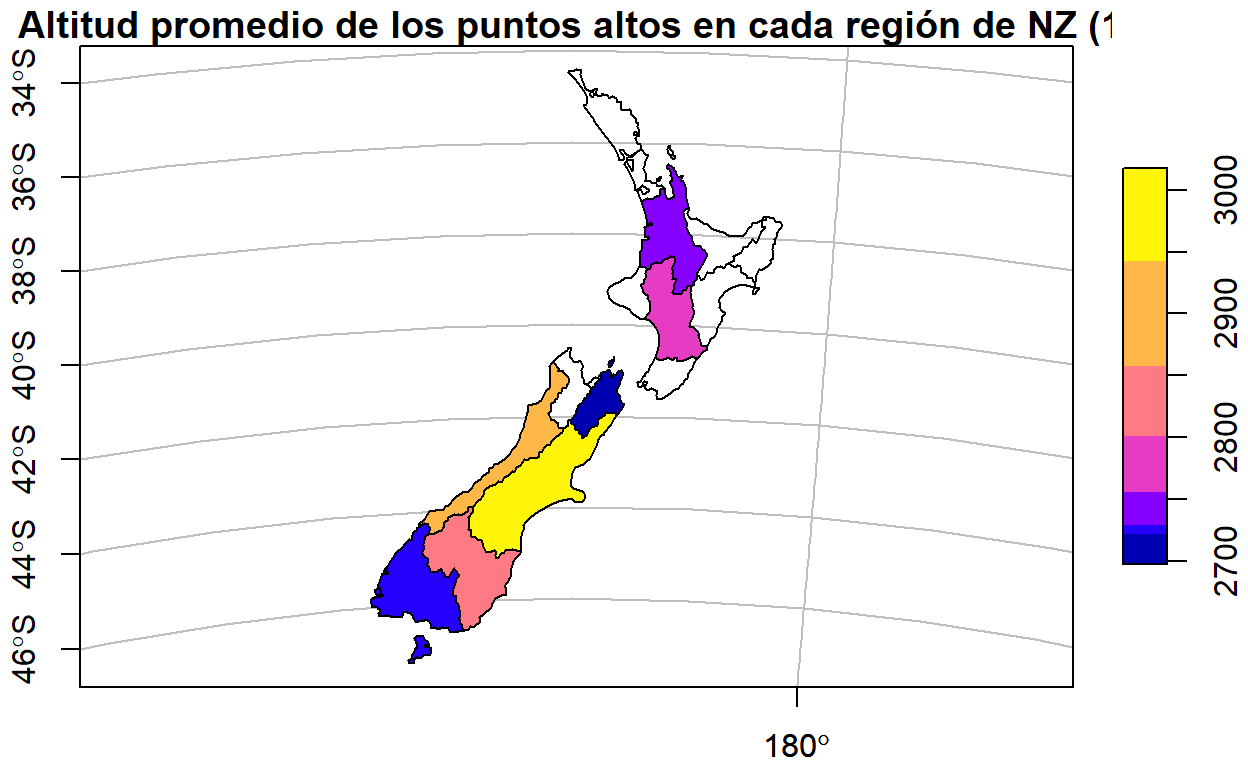
En el siguiente bloque de código, se utiliza summarize() para mostrar el promedio de altitud de los puntos más altos de Nueva Zelanda en cada región del país. Los datos de los puntos más altos están en el objeto nz_height y los de las regiones de Nueva Zelanda en el objeto nz.
# Promedio de altitud de puntos más altos para cada región de NZ
nz_altitud_promedio_x_region <-
nz_height %>%
aggregate(by = nz, FUN = mean)
# Mapa de altitud promedio en regiones
plot(
nz_altitud_promedio_x_region["elevation"],
main = "Altitud promedio de los puntos altos en cada región de NZ (1)",
axes = TRUE,
graticule = TRUE
)
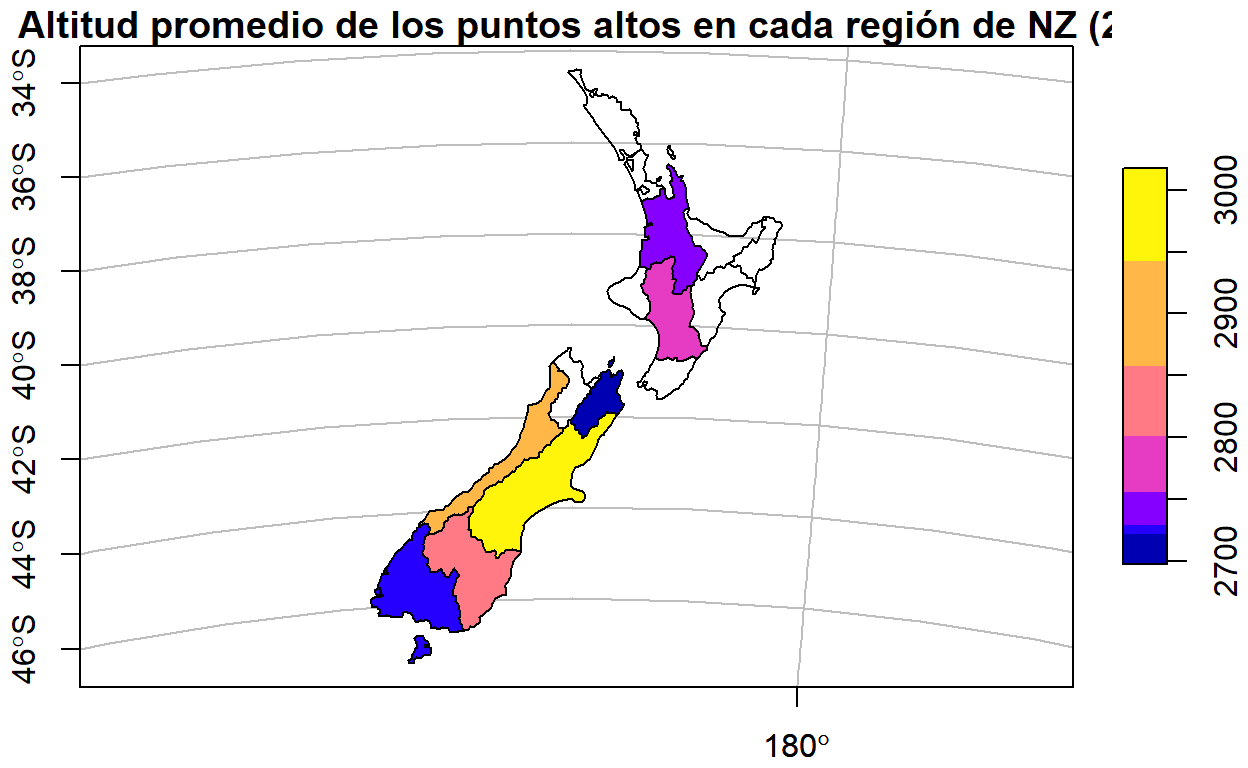
El siguiente bloque de código logra el mismo resultado, con los métodos de dplyr.
nz_altitud_promedio_x_region <-
nz %>%
st_join(nz_height) %>%
group_by(Name) %>%
summarize(elevation = mean(elevation))
# Mapa de altitud promedio en regiones
plot(
nz_altitud_promedio_x_region["elevation"],
main = "Altitud promedio de los puntos altos en cada región de NZ (2)",
axes = TRUE,
graticule = TRUE
)
Relaciones de distancia
La distancia entre dos objetos sf se calcula con el método st_distance(). Debe utilizarse un sistema espacial de referencia (SRS, CRS) con unidades apropiadas para la medición (ej. metros).
En el siguiente ejemplo, se calcula la distancia entre puntos correspondientes a los centroides de dos provincias. Antes, con el método st_transform(), la capa de provincias se transforma al CRS CRTM05, que utiliza metros como unidad de medición.
# Transformación de la capa de provincias al CRS CRTM05 (EPSG = 5367)
provincias_crtm05 <-
provincias %>%
st_transform(5367)
# Centroide de la provincia de San José
centroide_sanjose <-
provincias_crtm05 %>%
filter(provincia == "San José") %>%
st_centroid()
# Centroide la provincia de Alajuela
centroide_alajuela <-
provincias_crtm05 %>%
filter(provincia == "Alajuela") %>%
st_centroid()
# Distancia entre los centroides de San José y Alajuela
st_distance(centroide_sanjose, centroide_alajuela)
Units: [m]
[,1]
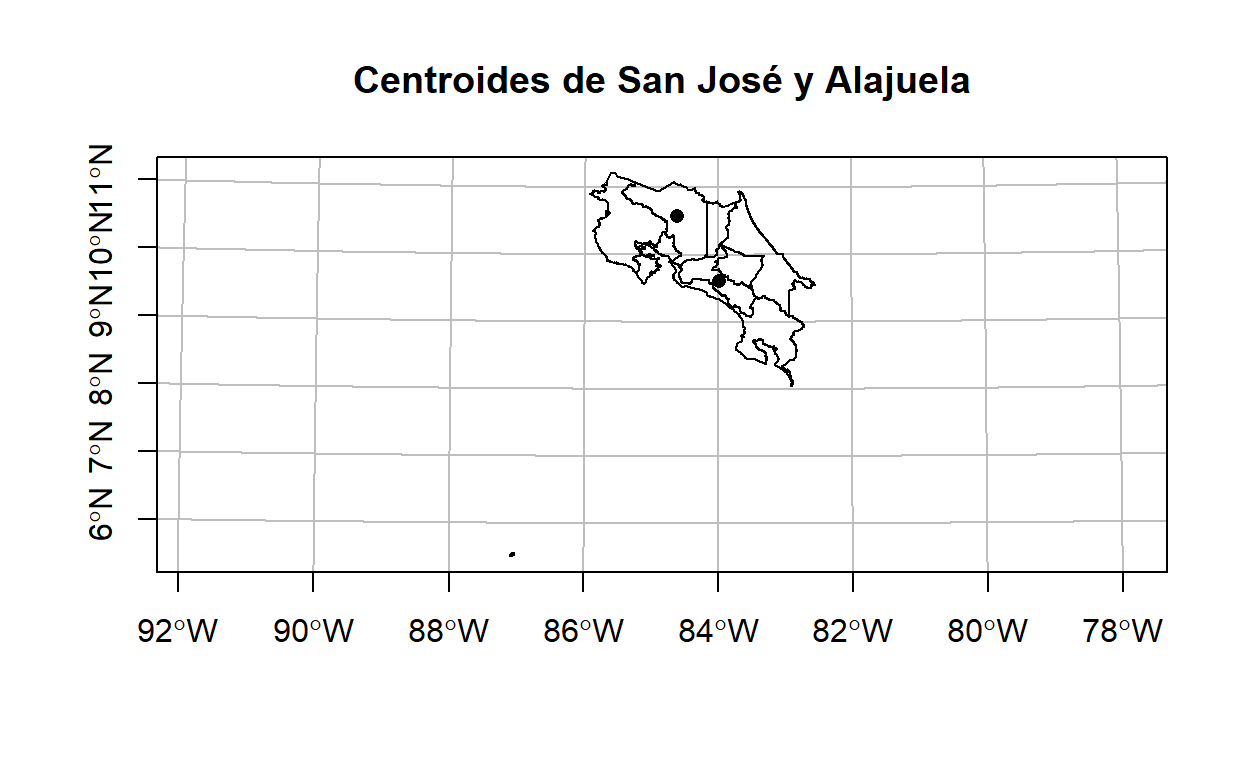
[1,] 126235.1# Mapa de los centroides
plot(
provincias_crtm05$geometry,
main = "Centroides de San José y Alajuela",
axes = TRUE,
graticule = TRUE,
reset = FALSE
)
plot(centroide_sanjose$geometry, pch = 16, add = TRUE)
plot(centroide_alajuela$geometry, pch = 16, add = TRUE)
Datos raster
Las operaciones espaciales en datos raster incluyen:
- Creación de subconjuntos espaciales (spatial subsetting).
- Álgebra de mapas (map algebra).
- Unión de capas raster (raster merging).
Manejo de datos espaciales con el paquete raster
En los siguientes ejemplos, se utilizarán los conjuntos de datos elev y grain que se crearon en la lección sobre datos de atributos, los cuales están también incluidos en el paquete spData.
Para mayor facilidad de manipulación y visualización, ambos conjuntos de datos pueden grabarse en disco.
# Especificación del directorio de trabajo (debe ser una ruta existente)
setwd("C:/Users/mfvargas/Downloads")
# Escritura de los objetos raster
writeRaster(elev, filename = "elev.asc", overwrite = TRUE)
writeRaster(grain, filename = "grain.asc", overwrite = TRUE)
Creación de subconjuntos espaciales
En la lección sobre operaciones con atributos, se explicó como recuperar subconjuntos de objetos raster, ya sea mediante su ID o su posición en filas y columnas. Los subconjuntos de objetos raster también pueden obtenerse mediante la especificación de coordenadas o de otros objetos espaciales.
# La función cellFromXY() retorna el ID de la celda correspondiente a una coordenada
id <- cellFromXY(elev, xy = c(0.1, 0.1))
elev[id]
[1] 16# El mismo resultado puede obtenerse con raster::extract()
raster::extract(elev, data.frame(x = 0.1, y = 0.1))
[1] 16También es posible consultar las celdas contenidas en la extensión (i.e. los límites) otro raster.
clip <- raster(
xmn = 0.9,
xmx = 1.8,
ymn = -0.45,
ymx = 0.45,
res = 0.3,
vals = rep(1, 9)
)
# Celdas de elev contenidas en la extensión de clip
elev[clip]
[1] 18 24[1] 18 24Los métodos explicados anteriormente solo retornan ID y valores de celdas. Con el operador [ y el argumento drop = FALSE pueden retornarse objetos raster.
# Objeto raster creado a partir de un rango de ID de celdas
elev[1:2, drop = FALSE]
class : RasterLayer
dimensions : 1, 2, 2 (nrow, ncol, ncell)
resolution : 0.5, 0.5 (x, y)
extent : -1.5, -0.5, 1, 1.5 (xmin, xmax, ymin, ymax)
crs : +proj=longlat +datum=WGS84 +ellps=WGS84 +towgs84=0,0,0
source : memory
names : layer
values : 1, 2 (min, max)# Objeto raster creado a partir de posiciones de filas y columnas
elev[1, 1:2, drop = FALSE]
class : RasterLayer
dimensions : 1, 2, 2 (nrow, ncol, ncell)
resolution : 0.5, 0.5 (x, y)
extent : -1.5, -0.5, 1, 1.5 (xmin, xmax, ymin, ymax)
crs : +proj=longlat +datum=WGS84 +ellps=WGS84 +towgs84=0,0,0
source : memory
names : layer
values : 1, 2 (min, max)Por último, también es posible obtener subconjuntos raster mediante la aplicación en un objeto raster de una “máscara” (mask) con la misma extensión y resolución, y que contenga valores lógicos o NA.
# Creación de una "máscara"
rmask = elev
values(rmask) = sample(c(NA, TRUE), 36, replace = TRUE)
# Creación de subconjuntos espaciales mediante la máscara
# Con el operador []
elev[rmask, drop = FALSE]
class : RasterLayer
dimensions : 6, 6, 36 (nrow, ncol, ncell)
resolution : 0.5, 0.5 (x, y)
extent : -1.5, 1.5, -1.5, 1.5 (xmin, xmax, ymin, ymax)
crs : +proj=longlat +datum=WGS84 +ellps=WGS84 +towgs84=0,0,0
source : memory
names : layer
values : 2, 31 (min, max)# Con la función mask()
mask(elev, rmask)
class : RasterLayer
dimensions : 6, 6, 36 (nrow, ncol, ncell)
resolution : 0.5, 0.5 (x, y)
extent : -1.5, 1.5, -1.5, 1.5 (xmin, xmax, ymin, ymax)
crs : +proj=longlat +datum=WGS84 +ellps=WGS84 +towgs84=0,0,0
source : memory
names : layer
values : 2, 31 (min, max)# Con la función overlay()
overlay(elev, rmask, fun = "max")
class : RasterLayer
dimensions : 6, 6, 36 (nrow, ncol, ncell)
resolution : 0.5, 0.5 (x, y)
extent : -1.5, 1.5, -1.5, 1.5 (xmin, xmax, ymin, ymax)
crs : +proj=longlat +datum=WGS84 +ellps=WGS84 +towgs84=0,0,0
source : memory
names : layer
values : 2, 31 (min, max)Ejemplos de creación de subconjuntos raster de la capa de altitud de Costa Rica
# Especificación del directorio de trabajo (debe ser una ruta existente)
setwd("C:/Users/mfvargas/Downloads")
# Capa vectorial de provincias de Costa Rica
provincias <-
st_read(
"https://raw.githubusercontent.com/gf0604-procesamientodatosgeograficos/2021i-datos/main/ign/delimitacion-territorial-administrativa/cr_provincias_simp_wgs84.geojson",
quiet = TRUE
)
# Obtención de la capa de altitud
alt <- getData(
"worldclim",
var = "alt",
res = .5,
lon = -84,
lat = 10
)
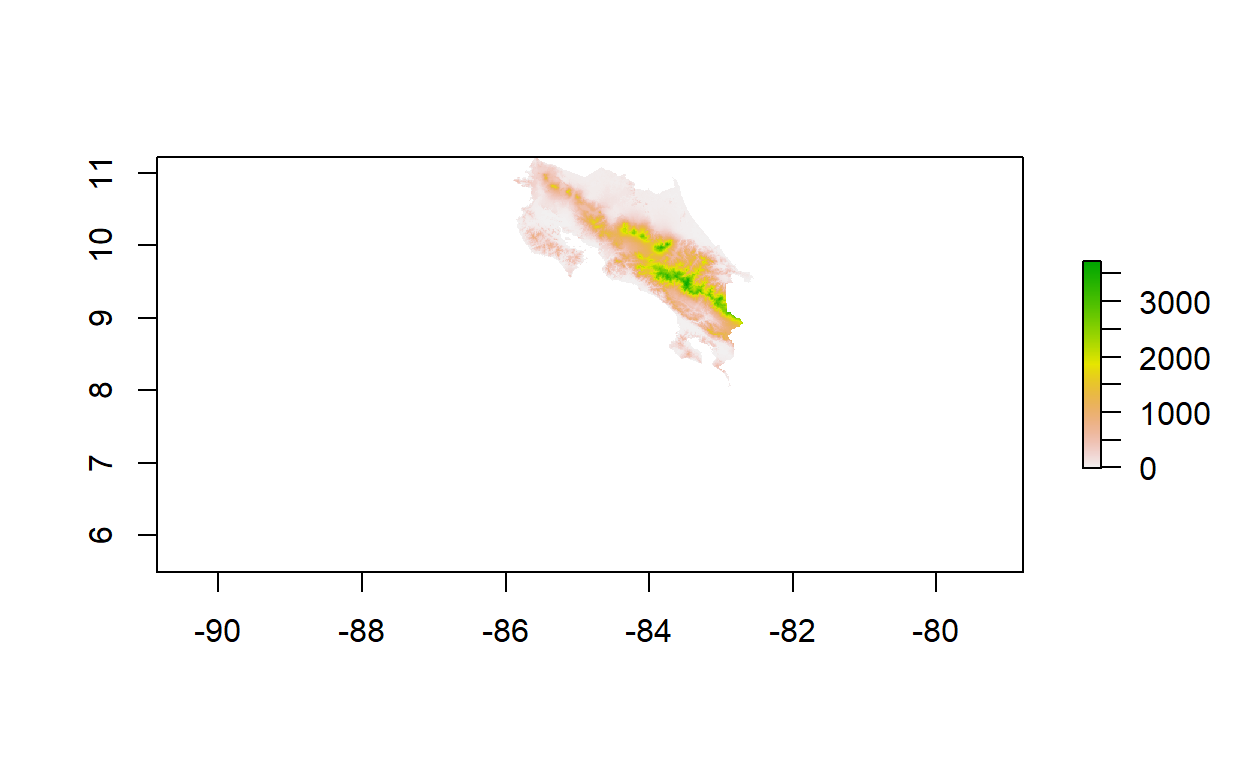
# Recorte de la capa de altitud
altitud_cr <-
alt %>%
mask(provincias) %>%
crop(provincias)
plot(altitud_cr)
# Altitud del punto (-84, 10)
altitud_cr[cellFromXY(altitud_cr, xy = c(-84, 10))]
[1] 1395# Altitud del Cerro Chirripó
altitud_cr[cellFromXY(altitud_cr, xy = c(-83.488667, 9.484083))]
[1] 3646# Altitud de la Catedral Metropolitana de San José
altitud_cr[cellFromXY(altitud_cr, xy = c(-84.078758, 9.932684))]
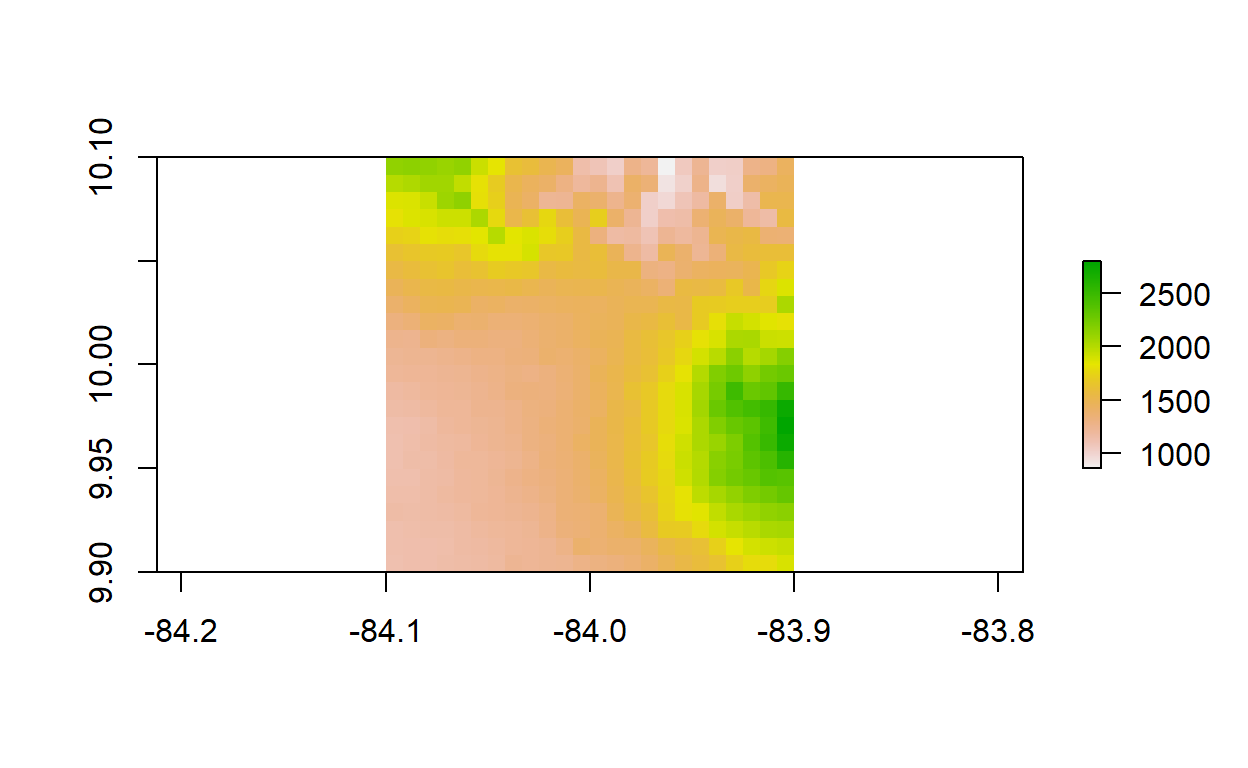
[1] 1152# Creación de un raster ubicado en el centro del país, alrededor de (-84, 10)
clip_centro_cr <-
raster(
xmn = -84.10,
xmx = -83.90,
ymn = 9.90,
ymx = 10.10,
res = 0.1
)
# Recorte de la capa de altitud con base en el raster del centro del país
altitud_centro_cr <- altitud_cr[clip_centro_cr, drop = FALSE]
plot(altitud_centro_cr)
# Mapeo con leaflet()
leaflet() %>%
setView(lng = -84.0, lat = 10.0, zoom = 7) %>%
addTiles(group = "OpenStreetMap") %>%
addRasterImage(altitud_cr, colors = "YlGnBu", group = "Altitud") %>%
addRasterImage(altitud_centro_cr, colors = "YlOrRd", group = "Altitud del centro del país") %>%
addLayersControl(
baseGroups = c("OpenStreetMap"),
overlayGroups = c("Altitud", "Altitud del centro del país")
)
Se recomienda el uso de las funciones calc() y overlay() sobre los operadores aritméticos, por ser más eficientes y manejar mejor conjuntos grandes.
Álgebra de mapas
El álgebra de mapas divide las operaciones raster en cuatro clases:
- Operaciones locales o de “celda por celda”.
- Operaciones focales o de “vecindario” (neighborhood). Generalmente, el valor de salida de cada celda proviene de un bloque de entrada de 3 x 3 celdas.
- Operaciones zonales. Son similares a las focales, pero el bloque de entrada puede tener tamaños y formas irregulares.
- Operaciones globales. Los valores de salida de cada celda provienen de uno o varios objetos raster completos.
Esta clasificación se basa en la cantidad o forma de las celdas utilizadas por cada pixel durante el procesamiento. Otras clasificaciones pueden estar basadas en el área de aplicación del análisis (ej. terreno, hidrología, teledetección).
Operaciones locales
Son operaciones realizadas celda por celda en una o varias capas raster.
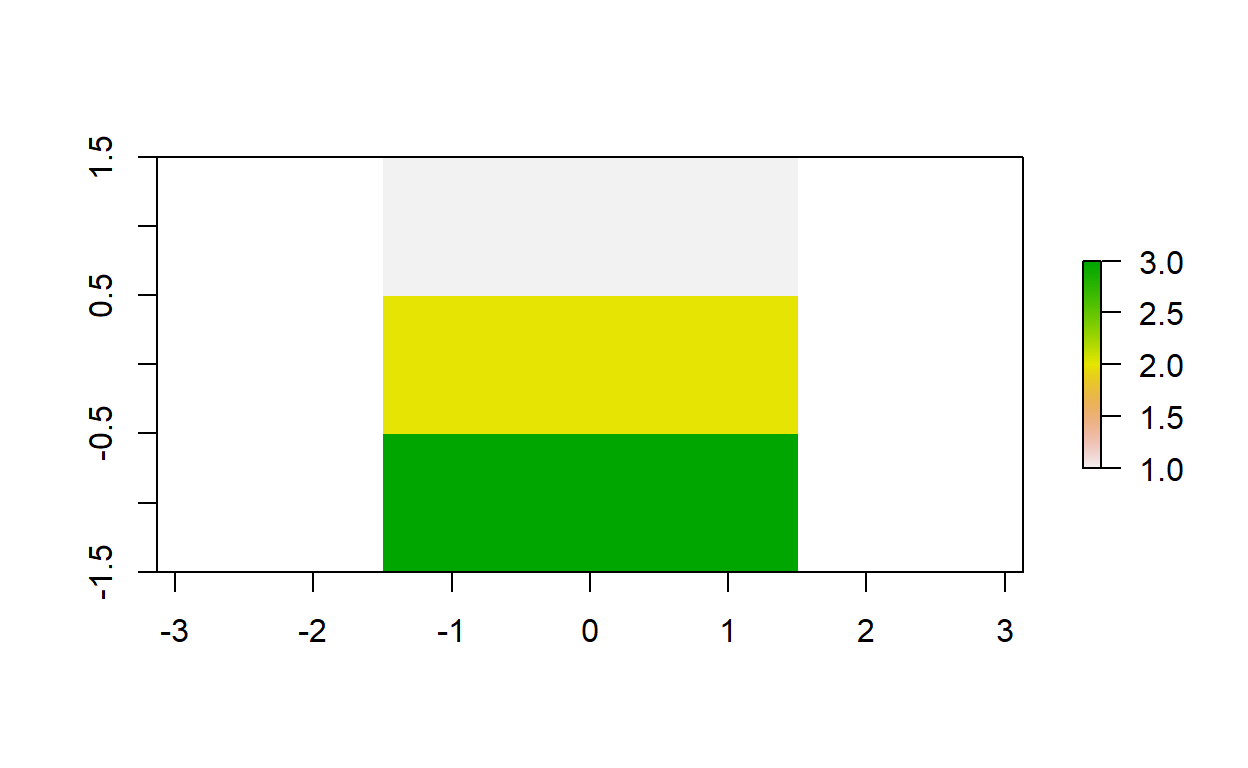
# Reclasificación de una capa raster
rcl <-
matrix(c(0, 12, 1, 12, 24, 2, 24, 36, 3), ncol = 3, byrow = TRUE)
recl <- reclassify(elev, rcl = rcl)
plot(recl)
# Álgebra raster con operadores aritméticos
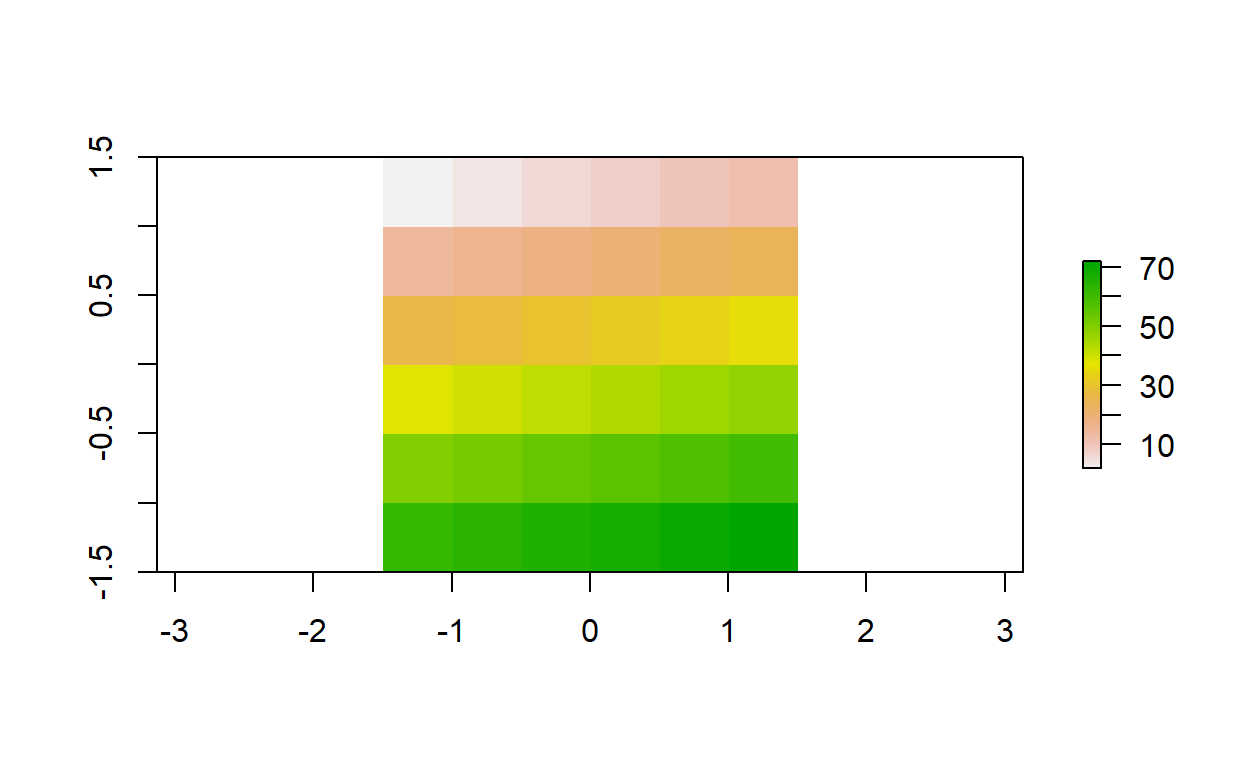
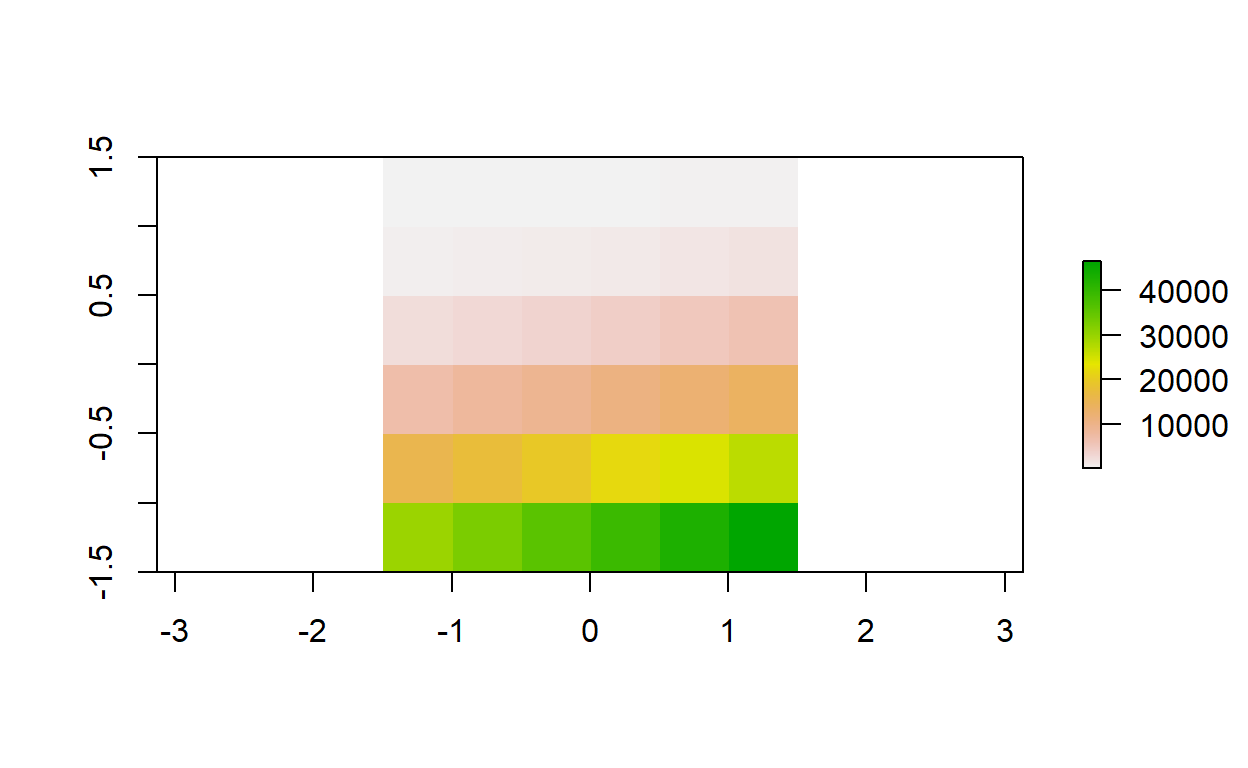
elev_doble <- elev + elev
plot(elev_doble)
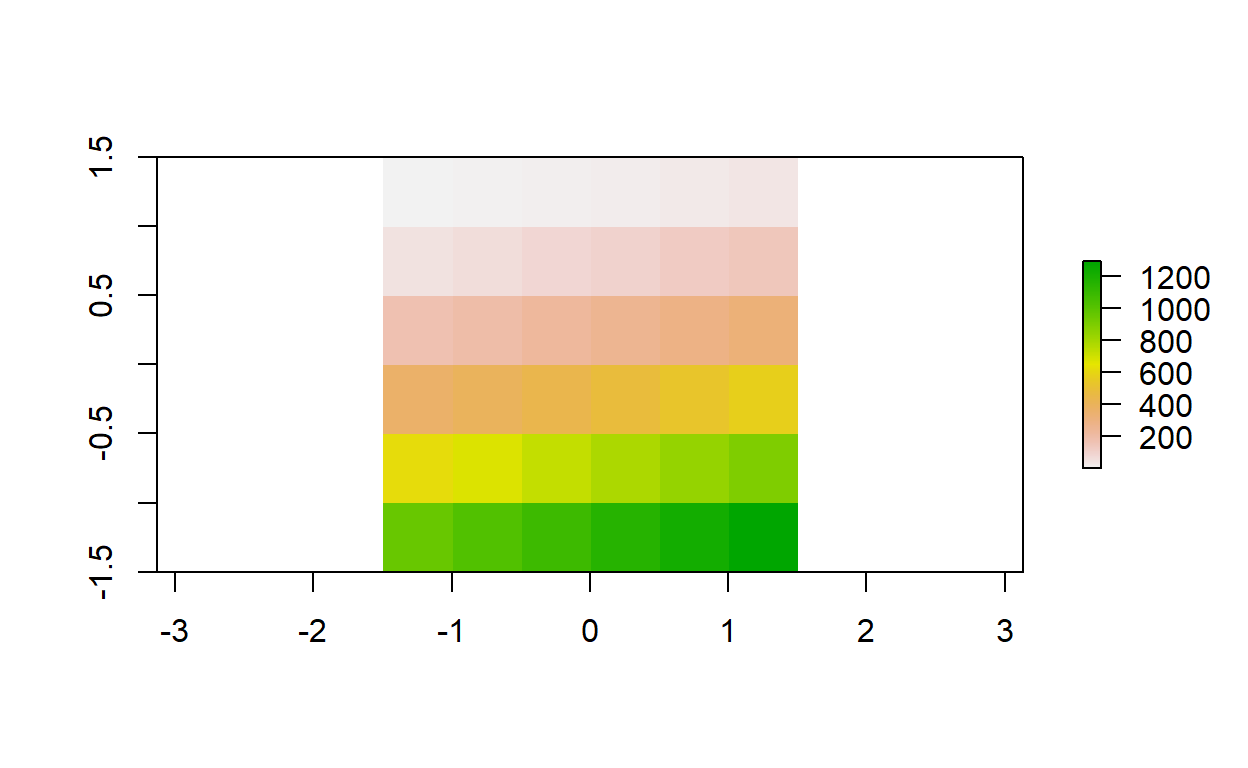
elev_cuadrado <- elev * elev
plot(elev_cuadrado)
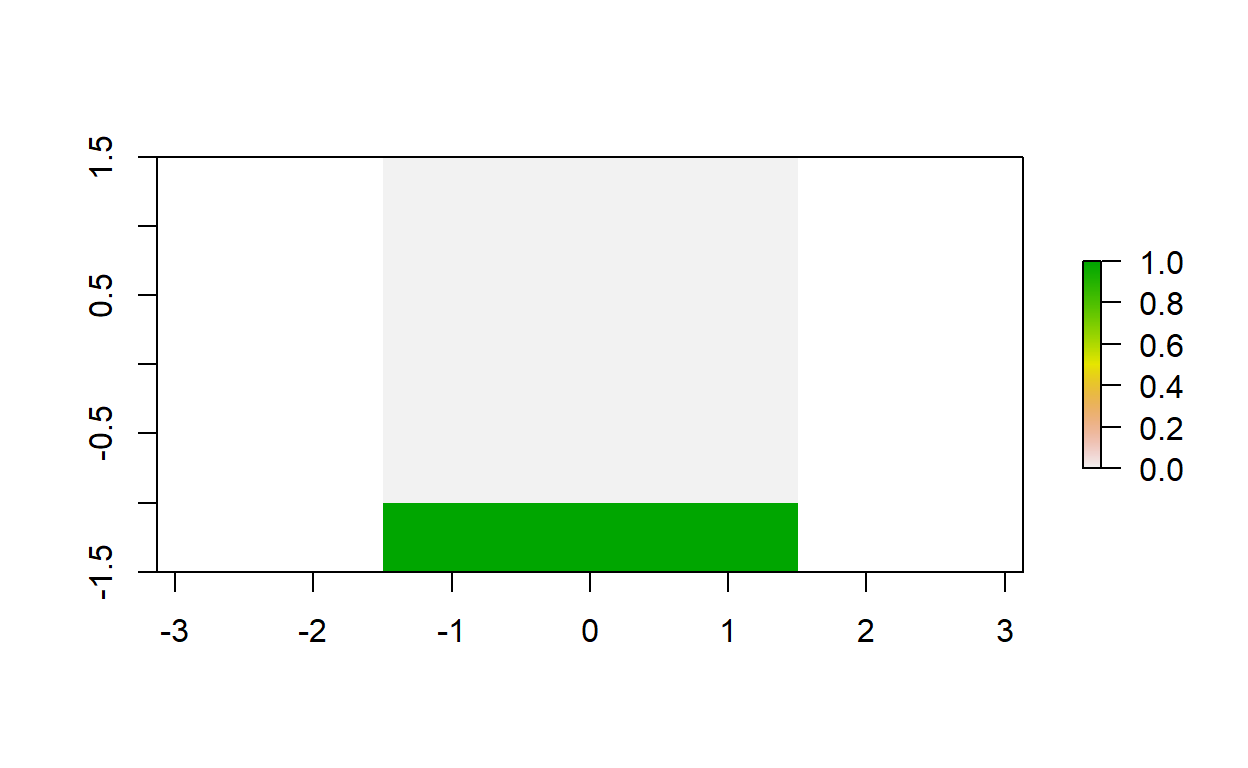
elev_mayor_30 <- elev > 30
plot(elev_mayor_30)
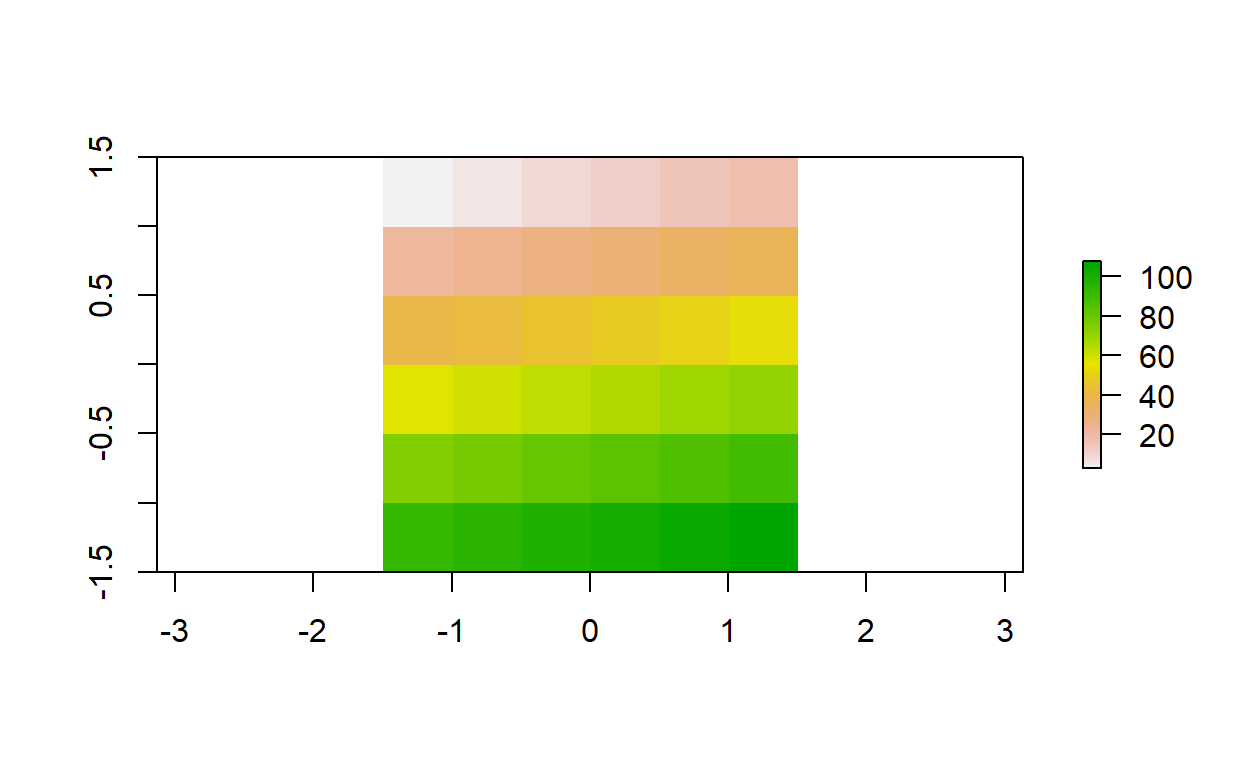
# Álgebra raster con calc()
elev_triple <- calc(
elev,
fun = function(x) {
x * 3
}
)
plot(elev_triple)
# Álgebra raster con overlay()
elev_cubo <- overlay(
elev,
fun = function(x) {
x ^ 3
}
)
plot(elev_cubo)
Operaciones focales
En este tipo de operaciones, el valor de salida de cada pixel procesado depende de un bloque compuesto por una celda central y sus vecinas. Este “vecindario” (también llamado kernel, filtro o “ventana móvil”) es típicamente de 3 x 3 celdas, pero puede tomar otras formas y tamaños. Una operación focal aplica una función de agregación (ej. promedio, mínimo, máximo) a todas las celdas del vecindario, utiliza la salida como nuevo valor de la celda central correspondiente y se mueve a la celda siguiente.
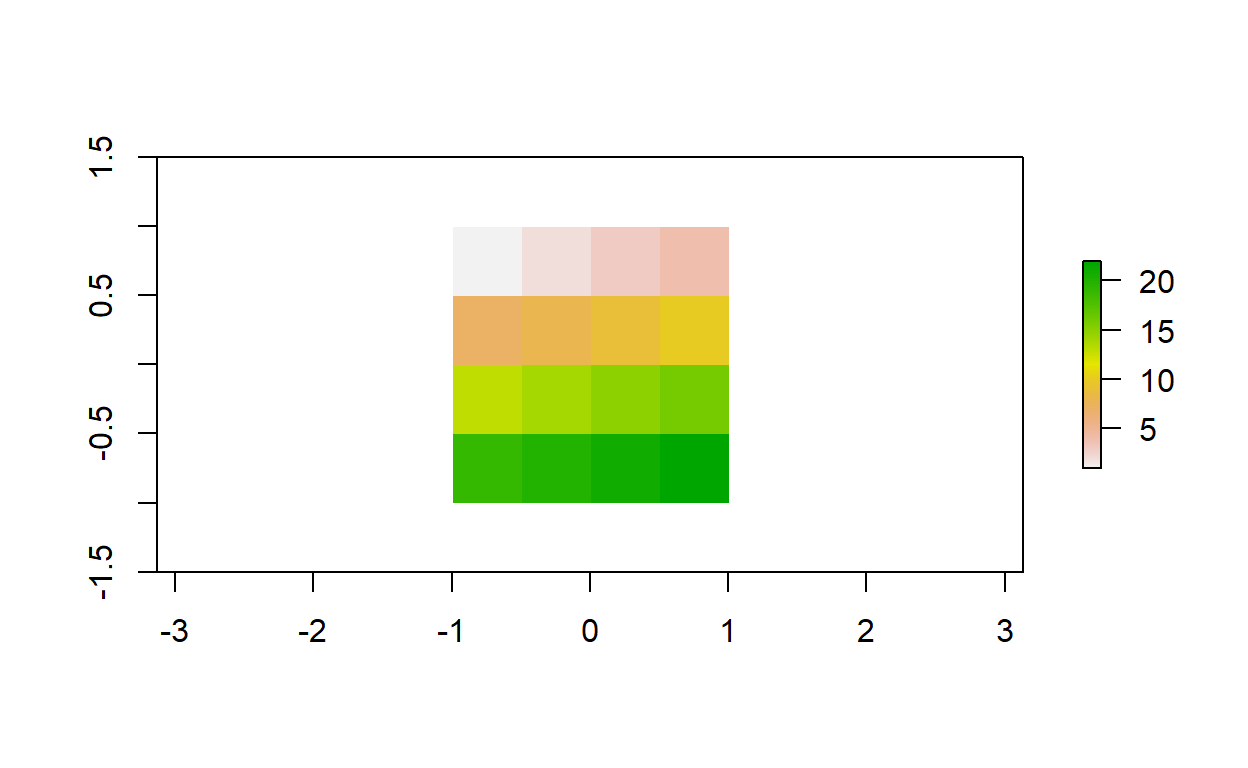
# Uso de focal() para obtener el valor mínimo en un vecindario de 3 x 3
r_focal <- focal(elev, w = matrix(1, nrow = 3, ncol = 3), fun = min)
plot(r_focal)
Las operaciones focales tienen aplicaciones en áreas como procesamiento de imágenes (ej. remoción de valores extremos) o análisis de terreno (ej. cálculo de pendiente o dirección de flujo).
Operaciones zonales
De manera similar a las focales, las operaciones zonales aplicación de agregación a múltiples celdas raster. Sin embargo, en el caso de las zonales, generalmente se usa un raster categórico que define las zonas, a diferencia de la ventana predefinida que se emplea en las focales. Por lo tanto, las celdas que definen el filtro zonal no deben ser necesariamente vecinas.
# Uso de zonal() para encontrar la elevación promedio de cada tipo de grano
zonal(elev, grain, fun = "mean") %>%
as.data.frame()
zone mean
1 1 17.75
2 2 18.50
3 3 19.25El siguiente bloque de código muestra como utilizar raster::extract() para calcular una estadística zonal con base en una capa vectorial.
# Cálculo de la altitud media en cada provincia
altitud_cr %>%
raster::extract(provincias["provincia"], fun = mean, na.rm = TRUE)
[,1]
[1,] 448.4330
[2,] 1138.0972
[3,] 596.6369
[4,] 1536.9147
[5,] 425.2522
[6,] 239.2353
[7,] 434.6728# Capa vectorial de provincias + columna de altitud media
provincias_altitud <-
provincias %>%
mutate(altitud_media = raster::extract(altitud_cr, provincias["provincia"], fun = mean, na.rm = TRUE))
# Mapeo
colores <-
colorNumeric(
palette = "YlGnBu",
domain = provincias_altitud$altitud_media,
na.color = "transparent"
)
leaflet() %>%
setView(lng = -84.0, lat = 10.0, zoom = 7) %>%
addTiles(group = "OpenStreetMap") %>%
addPolygons(
data = provincias_altitud,
fillColor = ~ colores(provincias_altitud$altitud_media),
fillOpacity = 0.7,
stroke = TRUE,
color = "black",
weight = 1,
popup = paste(
paste("<strong>Provincia:</strong>", provincias_altitud$provincia),
paste(
"<strong>Altitud media:</strong>",
round(provincias_altitud$altitud_media, digits = 2)
),
sep = '<br/>'
),
group = "Provincias - altitud media"
) %>%
addLayersControl(
baseGroups = c("OpenStreetMap"),
overlayGroups = c("Provincias - altitud media")
) %>%
addLegend(
position = "bottomleft",
pal = colores,
values = provincias_altitud$altitud_media,
group = "Provincias - altitud media",
title = "Altitud media"
)
Operaciones globales
Las operaciones globales pueden considerarse un caso particular de las operaciones zonales, en las cuales un raster completo corresponde a una zona. Las operaciones globales más comunes incluyen estadísticas descriptivas para todo un conjunto raster.
# Estadísticas descriptivas
cellStats(altitud_cr, mean)
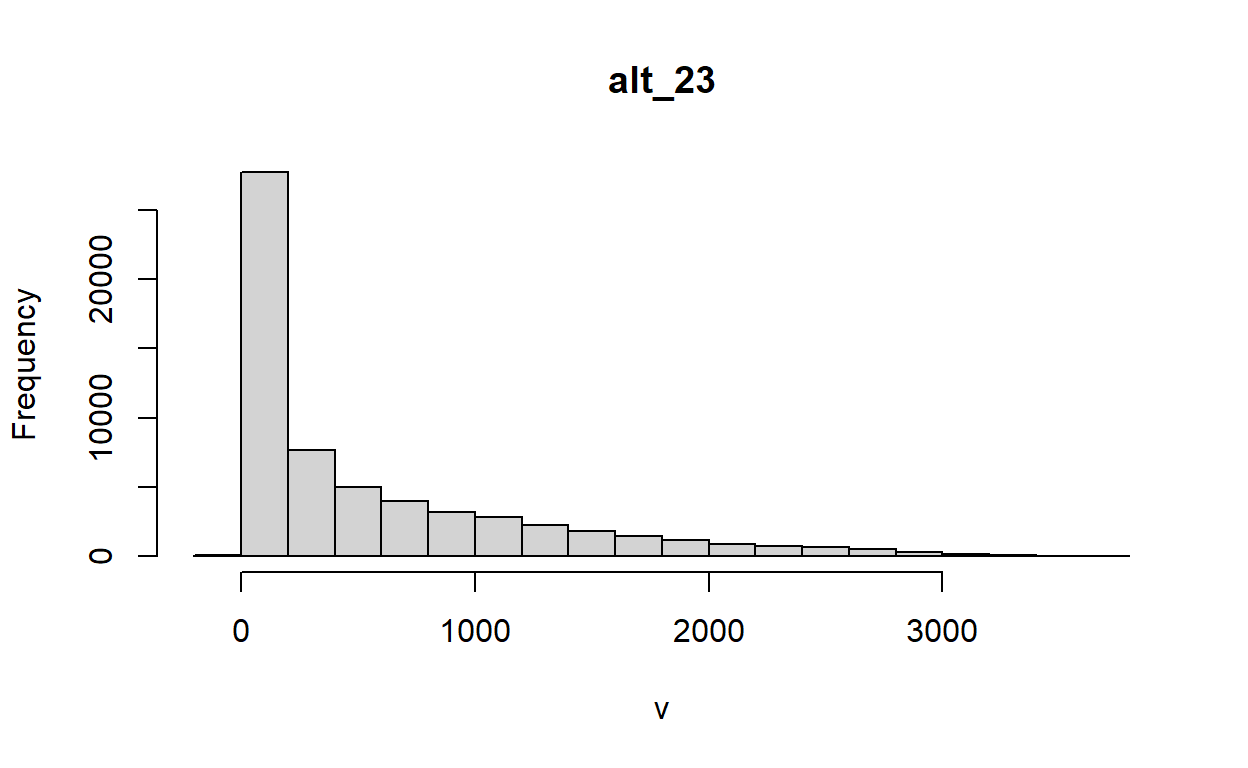
[1] 561.5629# Resumen
summary(altitud_cr)
alt_23
Min. -10
1st Qu. 59
Median 254
3rd Qu. 858
Max. 3732
NA's 314014# Histograma
hist(altitud_cr)
Otras operaciones globales incluyen cálculos de distancia y rasters de “peso” (weight rasters) (ej. distancia de cada celda a una celda objetivo, costo en combustible de cada celda a una celda objetivo).
Unión de capas raster
La función merge() une dos capas raster.
# Unión de los rasters de altitud de Haití y República Dominicana
altitud_hti <- getData("alt", country = "HTI", mask = TRUE)
altitud_dom <- getData("alt", country = "DOM", mask = TRUE)
altitud_hti_dom <- merge(altitud_hti, altitud_dom)
plot(altitud_hti_dom)
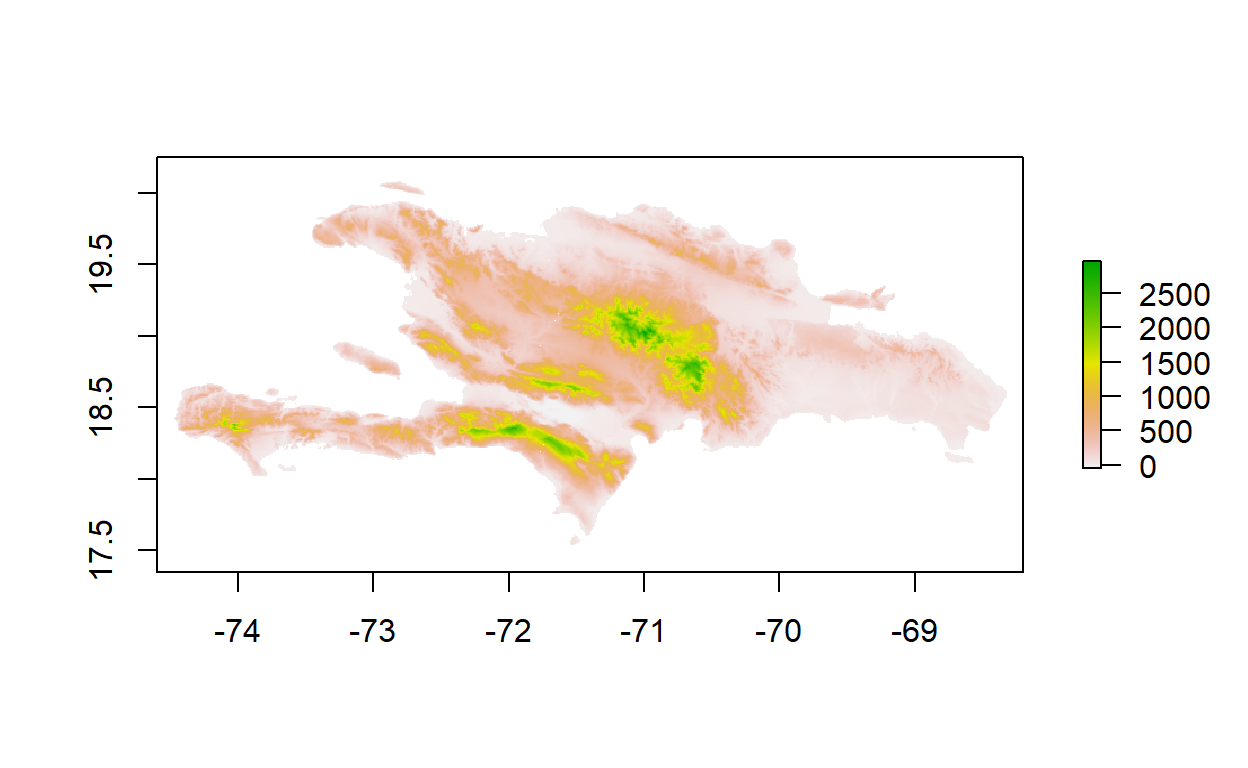
Si hay traslape entre las capas, se utiliza la del primer argumento de la función. El uso de esta función y de otras similares, como mosaic(), es frecuente, por ejemplo, en procesamiento de imágenes, cuando deben combinarse escenas tomadas en fechas diferentes.
Ejemplos de visualización
Se presentan diversas visualizaciones de los registros de presencia de la serpiente terciopelo (Bothrops asper) en Costa Rica, de acuerdo con los datos obtenidos a través de una consulta al portal de la Infraestructura Mundial de Información en Biodiversidad (GBIF).
Tabla
# Tabla de datos de registros de presencia
terciopelos %>%
st_drop_geometry() %>%
select(stateProvince,
canton,
locality,
year
) %>%
DT::datatable(
colnames = c("Provincia", "Cantón", "Localidad", "Año"),
rownames = FALSE,
options = list(
searchHighlight = TRUE
)
)
Gráfico
# Gráfico de registros de presencia por mes
terciopelos %>%
st_drop_geometry() %>%
group_by(mes = format(as.Date(eventDate, "%Y-%m-%d"), "%m")) %>%
summarize(suma_registros = n()) %>%
filter(!is.na(mes)) %>%
plot_ly(x = ~ mes,
y = ~ suma_registros) %>%
layout(title = "Registros de terciopelos (Bothrops asper) por mes",
xaxis = list(title = "Mes"),
yaxis = list(title = "Cantidad de registros"))
Mapa
# Mapa de registros de presencia
terciopelos %>%
select(stateProvince,
canton,
locality,
eventDate) %>%
leaflet() %>%
addProviderTiles(providers$OpenStreetMap.Mapnik, group = "OpenStreetMap") %>%
addProviderTiles(providers$Stamen.TonerLite, group = "Stamen Toner Lite") %>%
addProviderTiles(providers$Esri.WorldImagery, group = "Imágenes de ESRI") %>%
addCircleMarkers(
stroke = F,
radius = 4,
fillColor = 'green',
fillOpacity = 1,
popup = paste(
terciopelos$stateProvince,
terciopelos$canton,
terciopelos$locality,
terciopelos$eventDate,
sep = '<br/>'
),
group = "Terciopelos (Bothrops asper)"
) %>%
addLayersControl(
baseGroups = c("OpenStreetMap", "Stamen Toner Lite", "Imágenes de ESRI"),
overlayGroups = c("Terciopelos (Bothrops asper)")
) %>%
addMiniMap(
tiles = providers$Stamen.OpenStreetMap.Mapnik,
position = "bottomleft",
toggleDisplay = TRUE
)
Ejercicio de visualización
Utilizando R Markdown, genere un documento HTML para visualizar datos sobre el junco de los páramos (Junco vulcani) y publíquelo como un sitio web en GitHub Pages. Puede encontrar registros de presencia de la especie en el repositorio de datos del curso. Estos registros fueron obtenidos a través de una consulta al portal de la Infraestructura Mundial de Información en Biodiversidad (GBIF).
El sitio web debe contener visualizaciones en forma de:
- Tablas.
- Gráficos estadísticos.
- Mapas.