# Cargar bibliotecas
library(tidyverse)
library(DT)
library(sf)
library(terra)
library(tmap)13 Operaciones con datos espaciales
13.1 Resumen
Las operaciones espaciales, tales como las basadas en relaciones topológicas (ej. intersección, traslape, contención, cobertura), en el caso de los datos vectoriales, y el álgebra de mapas, en el caso de los datos raster, constituyen un componente esencial del procesamiento de datos geoespaciales. Varias de las operaciones con datos de atributos estudiadas en el capítulo anterior tienen contrapartes espaciales.
Las operaciones espaciales para datos vectoriales incluyen creación de subconjuntos espaciales, unión de datos espaciales, agregación de datos espaciales y relaciones de distancia, entre otras. Por su parte, las operaciones espaciales para datos raster incluyen creación de subconjuntos espaciales y álgebra de mapas, entre otras.
13.2 Trabajo previo
13.2.1 Lecturas
Lovelace, R., Nowosad, J., & Münchow, J. (2019). Geocomputation with R (capítulo 4). CRC Press. https://geocompr.robinlovelace.net/
13.3 Carga de bibliotecas
13.4 Carga de datos
13.4.1 Cantones de Costa Rica
Este archivo proviene de un geoservicio de tipo Web Feature Service (WFS) publicado por el Instituto Geográfico Nacional (IGN). Las geometrías se simplificaron para reducir el tamaño del archivo.
Archivo GPKG de cantones de Costa Rica para mayo de 2025 (con geometrías simplificadas)
Código para cargar datos de cantones
# Cargar datos de cantones de Costa Rica
cantones <- st_read(
"https://github.com/gf0604-procesamientodatosgeograficos/2025-i/raw/refs/heads/main/datos/ign/cantones-simplificados.gpkg",
quiet = TRUE
)13.4.2 Registros de presencia de félidos de Costa Rica
Este archivo, con registros de presencia de especies silvestres de la familia Felidae en Costa Rica, proviene de una consulta al portal de datos de la Infraestructura Mundial de Información en Biodiversidad (GBIF).
Archivo CSV de registros de presencia de félidos silvestres de Costa Rica
Código para cargar datos de félidos
# Lectura de un archivo CSV con registros de presencia de félidos en Costa Rica
felidos <- st_read(
"https://raw.githubusercontent.com/gf0604-procesamientodatosgeograficos/2025-i/refs/heads/main/datos/gbif/felidos.csv",
options = c(
"X_POSSIBLE_NAMES=decimalLongitude", # columna de longitud decimal
"Y_POSSIBLE_NAMES=decimalLatitude" # columna de latitud decimal
),
quiet = TRUE
)
# Asignar CRS WGS84
st_crs(felidos) <- 4326Código para generar el mapa de cantones y registros de presencia de félidos
# Crear columna con el enlace a los detalles del registro original
felidos <- felidos |>
mutate(
registro = paste0(
"<a href='", occurrenceID,
"' target='_blank'>Detalle del registro</a>"
)
)
# Especificar el modo interactivo
tmap_mode("view")
# Definir el mapa de cantones y registros de félidos
mapa_cantones_felidos <-
# Centro y nivel inicial de zoom
tm_view(
set_view = c(lon = -84.2, lat = 9.6, zoom = 7)
) +
# Capas base
tm_basemap(c("OpenStreetMap", "Esri.WorldImagery")) +
# Capa de cantones (polígonos)
tm_shape(cantones, name = "Cantones") +
tm_borders() +
# Capa de félidos (puntos)
tm_shape(felidos, name = "Félidos") +
tm_dots(
fill = "species",
fill.scale = tm_scale(values = "brewer.set1"), # paleta Set1
fill.legend = tm_legend(title = "Especie"), # título de la leyenda
# Otras propiedades visuales
size = 0.5,
id = "species",
popup.vars = c(
"Localidad" = "locality",
"Fecha" = "eventDate",
"Fuente" = "institutionCode",
"Registro" = "registro" # enlace al detalle del registro de presencia
),
popup.format = list(html.escape = FALSE) # para hacer "clicables" los enlaces HTML
) +
# Definir la escala gráfica
tm_scalebar(position = c("left", "bottom"))
# Mostrar el mapa
mapa_cantones_felidosMapa de cantones y registros de presencia de félidos de Costa Rica
13.5 Introducción
Este capítulo brinda una visión general de las operaciones espaciales para datos vectoriales implementadas en el paquete sf, y para datos raster implementadas en el paquete terra.
13.6 Datos vectoriales
Las operaciones espaciales para datos vectoriales incluyen:
- Creación de subconjuntos espaciales (spatial subsetting).
- Unión de datos espaciales (spatial joining).
- Agregación de datos espaciales (spatial aggregation).
- Relaciones de distancia.
Seguidamente, se explica como maneja estas operaciones el paquete sf. Antes, se cubre el tema de las relaciones topológicas, el cual es importante en varias operaciones espaciales.
13.6.1 Relaciones topológicas
Las relaciones topológicas o relaciones topológicas binarias son expresiones lógicas (verdaderas o falsas) sobre las relaciones espaciales entre dos objetos. Por ejemplo, si a y b son dos objetos espaciales (ej. puntos, líneas, polígonos), se pueden considerar relaciones topológicas como las siguientes:
a interseca a b
a es adyacente a b
a está dentro de b
b está contenido en a
Los diferentes tipos de relaciones topológicas están estandarizados en el modelo DE-9IM (Dimensionally Extended 9-Intersection Model) y se ilustran en la Figura 13.1. Esta figura muestra también los métodos del paquete sf que implementan las relaciones topológicas (ej. st_intersects(), st_overlaps(), st_winthin(), st_contains()).
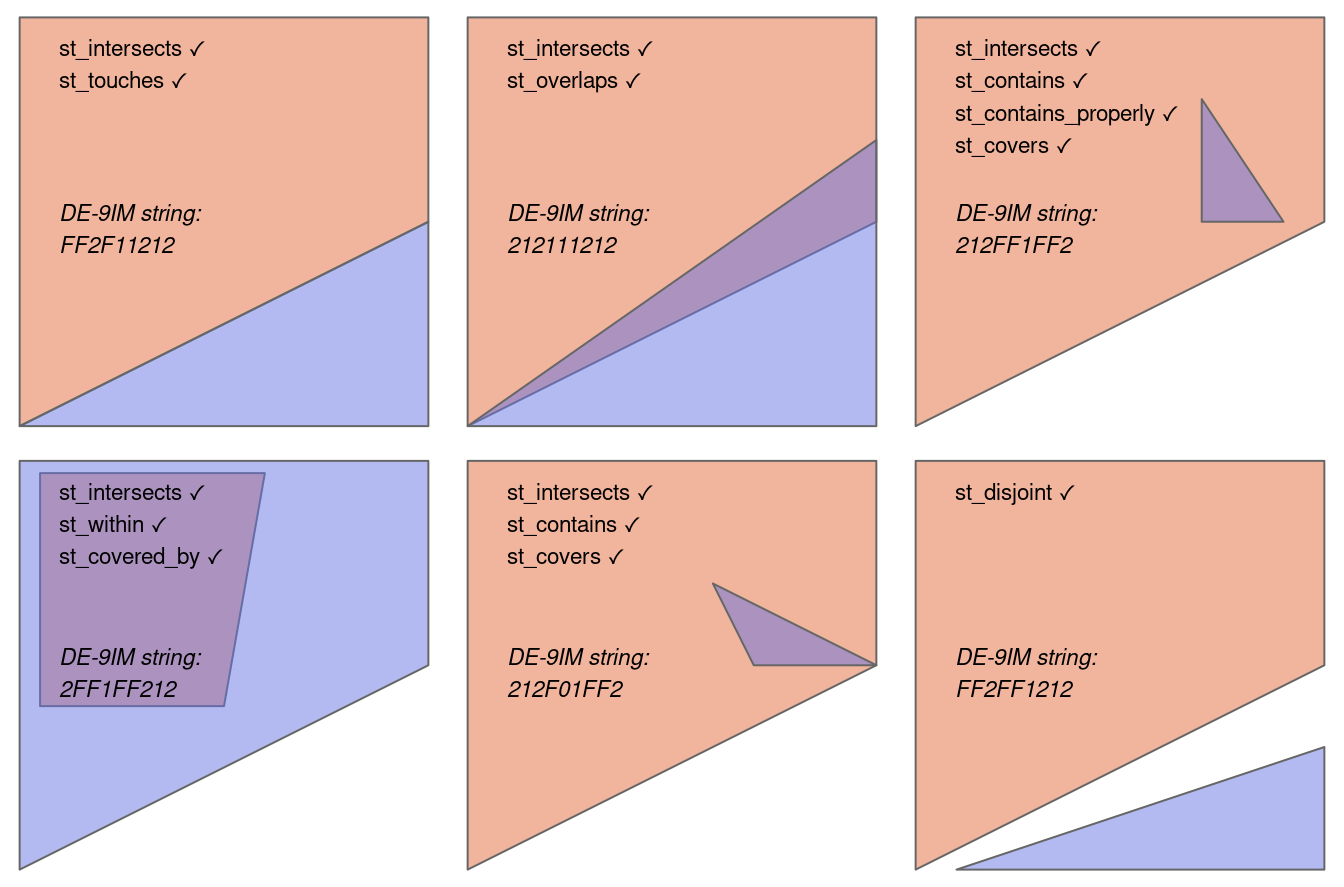
Nótese que para algunas relaciones topológicas (ej. intersección, traslape), el orden no es importante, mientras que para otras sí lo es (ej. contención).
A las relaciones topológicas se les conoce también como predicados espaciales y así se les denomina en la documentación de sf.
13.6.2 Manejo de datos espaciales con el paquete sf
13.6.2.1 Creación de subconjuntos espaciales
La creación de subconjuntos espaciales consiste en retornar objetos espaciales que cumplen con algún predicado espacial. Es análoga a la creación de subconjuntos por datos de atributos. Puede realizarse a través de los operadores [ y $ del paquete base de R o por medio de la función filter() de dplyr. Esta última opción es la que se usa en los siguiente ejemplos.
En este caso, la función filter() usa tres argumentos: x, y y .predicate, en donde x e y son dos conjuntos de datos espaciales y .predicate el predicado espacial que los relaciona.
13.6.2.1.1 Ejemplo: registros de presencia de félidos en el cantón Sarapiquí y sus alrededores
El siguiente bloque de código crea dos subconjuntos espaciales del conjunto de registros de presencia de félidos, al relacionarlo con el polígono del cantón Sarapiquí mediante los predicados espaciales st_within() y st_is_within_distance().
Código
# Polígono del cantón de Sarapiquí
sarapiqui <- filter(cantones, CANTÓN == "Sarapiquí")
# Puntos de félidos ubicados dentro del cantón Sarapiquí
felidos_dentro_sarapiqui <- st_filter(
x = felidos,
y = sarapiqui,
.predicate = st_within
)
# Puntos de félidos ubicados dentro de una distancia
# de hasta 10 km del cantón Sarapiquí
felidos_10km_sarapiqui <- st_filter(
x = felidos,
y = sarapiqui,
.predicate = function(a, b) st_is_within_distance(a, b, 10000)
)Los subconjuntos espaciales creados se despliegan en los mapas que se muestran seguidamente.
Código
# Especificar el modo interactivo
tmap_mode("view")
# Definir el mapa
mapa_felidos_dentro_sarapiqui <-
# Capas base
tm_basemap(c("OpenStreetMap", "Esri.WorldImagery")) +
# Capa de Sarapiquí (polígonos)
tm_shape(sarapiqui, name = "Sarapiquí") +
tm_borders() +
# Capa de félidos (puntos)
tm_shape(felidos_dentro_sarapiqui, name = "Félidos") +
tm_dots(
fill = "species",
fill.scale = tm_scale(values = "brewer.set1"),
fill.legend = tm_legend(title = "Especie"),
# Otras propiedades visuales
size = 0.5,
id = "species",
popup.vars = c(
"Localidad" = "locality",
"Fecha" = "eventDate",
"Fuente" = "institutionCode",
"Registro" = "registro"
),
popup.format = list(html.escape = FALSE)
) +
# Definir la escala gráfica
tm_scalebar(position = c("left", "bottom"))
# Mostrar el mapa
mapa_felidos_dentro_sarapiqui
# Especificar el modo interactivo
tmap_mode("view")
# Definir el mapa
mapa_felidos_10km_sarapiqui <-
# Capas base
tm_basemap(c("OpenStreetMap", "Esri.WorldImagery")) +
# Capa de Sarapiquí (polígonos)
tm_shape(sarapiqui, name = "Sarapiquí") +
tm_borders() +
# Capa de félidos (puntos)
tm_shape(felidos_10km_sarapiqui, name = "Félidos") +
tm_dots(
fill = "species",
fill.scale = tm_scale(values = "brewer.set1"),
fill.legend = tm_legend(title = "Especie"),
# Otras propiedades visuales
size = 0.5,
id = "species",
popup.vars = c(
"Localidad" = "locality",
"Fecha" = "eventDate",
"Fuente" = "institutionCode",
"Registro" = "registro"
),
popup.format = list(html.escape = FALSE)
) +
# Definir la escala gráfica
tm_scalebar(position = c("left", "bottom"))
# Mostrar el mapa
mapa_felidos_10km_sarapiquiMapa de registros de presencia de félidos localizados dentro del cantón Sarapiquí.
Mapa de registros de presencia de félidos localizados dentro de una distancia de hasta 10 km del cantón Sarapiquí.
13.6.2.2 Unión (join) de datos espaciales
Como se estudió en el capítulo sobre operaciones de datos de atributos, la unión (join) “no espacial” de dos conjuntos de datos se basa en una o varias columnas (llamadas llaves o keys) que están presentes en ambos conjuntos. Las uniones espaciales se basan en un principio similar pero, en lugar de columnas, la relación entre los conjuntos se realiza a través de un predicado espacial.
En sf, la unión de datos espaciales se implementa a través de la función st_join(), la cual tiene tres argumentos principales: x, y y join, en donde x e y son dos conjuntos de datos espaciales y join un predicado espacial que, por defecto, es st_intersects. st_join() agrega una o varias columnas a x, provenientes de y, para las filas que satisfacen el predicado.
13.6.2.2.1 Ejemplo: riqueza de especies de félidos en los cantones de Costa Rica
Los siguientes bloques de código calculan la riqueza de especies de félidos en los cantones de Costa Rica, esto es, la cantidad de especies en cada cantón.
El proceso se divide en los siguientes pasos:
- Unión espacial de félidos y cantones (esto le agrega a cada registros de félidos el código de cantón correspondiente a su ubicación).
- Conteo de la cantidad de especies de félidos en cada cantón (por código de cantón).
- Unión no espacial de cantones con el dataframe con el conteo de especies en cantones (esto le agrega a cada cantón la cantidad de especies de félidos).
- Generación del mapa de riqueza de especies.
1. Unión espacial de félidos y cantones
Con st_join(), al conjunto de registros de presencia de félidos, se le agrega la columna cod_canton del conjunto de cantones, mediante el predicado espacial st_within().
Código
# Unión espacial de félidos y cantones (solo la columna CÓDIGO_CANTÓN),
# mediante el predicado st_within().
# Como resultado, CÓDIGO_CANTÓN se une al conjunto de datos de félidos.
felidos_union_cantones <- st_join(
x = felidos,
y = dplyr::select(cantones, CÓDIGO_CANTÓN), # selección de columna CÓDIGO_CANTÓN
join = st_within
)
# Despliegue de los datos unidos de félidos y la columna CÓDIGO_CANTÓN de cantones
felidos_union_cantones |>
st_drop_geometry() |>
dplyr::select(species, locality, CÓDIGO_CANTÓN) |>
datatable(
colnames = c("Especie", "Localidad", "Código de cantón"),
options = list(
pageLength = 5,
language = list(url = '//cdn.datatables.net/plug-ins/1.10.11/i18n/Spanish.json')
)
)2. Conteo de la cantidad de especies de félidos en cada cantón
Se realiza una agrupación por código de cantón con group_by(cod_canton) y se crea la columna riqueza_especies_felidos con summarize(riqueza_especies_felidos = n_distinct(species, na.rm = TRUE)). La función n_distinct() cuenta la cantidad de valores diferentes de una columna; en este caso, la cantidad de especies en cada cantón.
Código
# Conteo de la cantidad de especies de félidos en cantones
riqueza_especies_felidos_cantones <-
felidos_union_cantones |>
st_drop_geometry() |>
group_by(CÓDIGO_CANTÓN) |>
summarize(riqueza_especies_felidos = n_distinct(species, na.rm = TRUE))
# Despliegue de la cantidad de especies de félidos en cada cantón
riqueza_especies_felidos_cantones |>
arrange(desc(riqueza_especies_felidos)) |>
datatable(
colnames = c("Código de cantón", "Riqueza de especies de félidos"),
options = list(
pageLength = 5,
language = list(url = '//cdn.datatables.net/plug-ins/1.10.11/i18n/Spanish.json')
)
)Nótese que hay registros que no están dentro del polígono de ningún cantón y tienen un valor NA en cod_canton. Esto puede deberse a errores de georreferenciación o a políticas de los publicadores de datos para no revelar la ubicación exacta de especies amenazadas.
3. Unión no espacial de cantones con el dataframe de riqueza de especies en cantones
Con left_join(), al conjunto de datos de cantones (incluyendo sus geometrías), se le agrega la columna riqueza_especies_felidos, con el conteo de especies por cantón. La función replace_na() se utiliza para reemplazar con 0 los registros de riqueza_especies_felidos que contienen NA.
Código
# Unión (no espacial) de cantones y riqueza de especies
cantones_union_riqueza <-
left_join(
x = cantones,
y = dplyr::select(riqueza_especies_felidos_cantones, CÓDIGO_CANTÓN, riqueza_especies_felidos),
by = "CÓDIGO_CANTÓN"
) |>
replace_na(list(riqueza_especies_felidos = 0))
# Despliegue de los datos de riqueza de especies en cantones
cantones_union_riqueza |>
st_drop_geometry() |>
dplyr::select(CANTÓN, riqueza_especies_felidos) |>
arrange(desc(riqueza_especies_felidos)) |>
datatable(
colnames = c("Cantón", "Riqueza de especies de félidos"),
options = list(
pageLength = 5,
language = list(url = '//cdn.datatables.net/plug-ins/1.10.11/i18n/Spanish.json')
)
)4. Generación del mapa de riqueza de especies
Con las geometrías de cantones y la riqueza de especies en el mismo conjunto de datos, puede generarse un mapa de coropletas.
Código para generar el mapa de riqueza de especies de félidos en cantones
# Activar el modo interactivo
tmap_mode("view")
# Definir el mapa
mapa_riqueza_felidos_cantones <-
tm_view(
set_view = c(lon = -84.2, lat = 9.6, zoom = 7)
) +
tm_basemap("OpenStreetMap", "Esri.WorldImagery") +
tm_shape(cantones_union_riqueza, name = "Riqueza de especies de félidos") +
tm_fill(
fill = "riqueza_especies_felidos",
fill.scale = tm_scale_intervals(
style = "quantile",
values = "Reds"
),
fill.legend = tm_legend(title = "Riqueza"),
id = "CANTÓN",
popup.vars = c(
"Cantón" = "CANTÓN",
"Riqueza" = "riqueza_especies_felidos"
)
) +
tm_borders(col = "black", lwd = 0.5) +
tm_scale_bar(position = c("left", "bottom"))
# Mostrar el mapa
mapa_riqueza_felidos_cantonesMapa de riqueza de especies de félidos en cantones de Costa Rica
13.6.3 Ejercicios
- Elabore un mapa de riqueza de especies de vipéridos en las provincias de Costa Rica. Impleméntelo como un mapa de coropletas en Leaflet.
Utilice las siguientes fuentes de datos:
13.7 Datos raster
Las operaciones espaciales para datos raster incluyen:
- Creación de subconjuntos espaciales (spatial subsetting).
- Álgebra de mapas (map algebra).
13.7.1 Manejo de datos espaciales con el paquete terra
13.7.1.1 Creación de subconjuntos espaciales
Para ejemplificar las operaciones, se utilizarán datos de temperatura media anual de WorldClim para Costa Rica.
# Cargar datos de temperatura media anual
temperatura <- rast(
"https://github.com/gf0604-procesamientodatosgeograficos/2025-i/raw/refs/heads/main/datos/worldclim/temperatura.tif"
)
# Desplegar datos
plot(temperatura)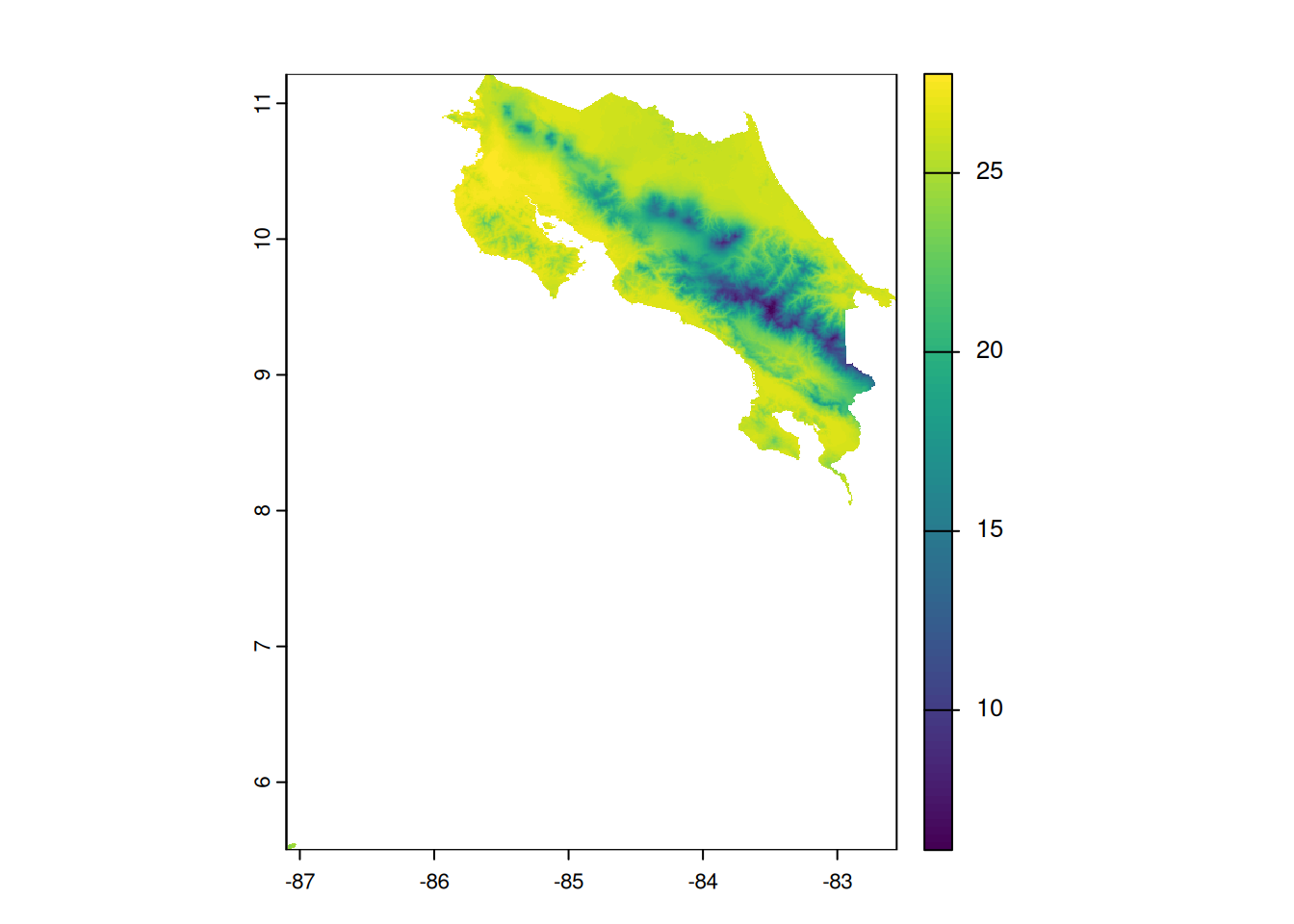
El método extract() extrae valores de un objeto raster.
# Temperatura media anual del Cerro Chirripó
extract(
temperatura,
cbind(-83.488667, 9.484083) # matriz de 1 fila (x, y)
) temperatura
1 6.366667# Temperatura media anual en la Catedral Metropolitana de San José
extract(
temperatura,
cbind(-84.078758, 9.932684)
) temperatura
1 20.86667El método crop() recorta un objeto SpatRaster de acuerdo con el contorno de otro objeto espacial, raster o vectorial.
El siguiente bloque de código recorta la capa de temperatura media anual de Costa Rica de acuerdo con el objeto vectorial del cantón Sarapiquí.
# Recorte de la capa de temperatura
temperatura_sarapiqui <-
temperatura |>
crop(sarapiqui) |>
mask(sarapiqui)
# Despliegue de la capa recortada
plot(temperatura_sarapiqui)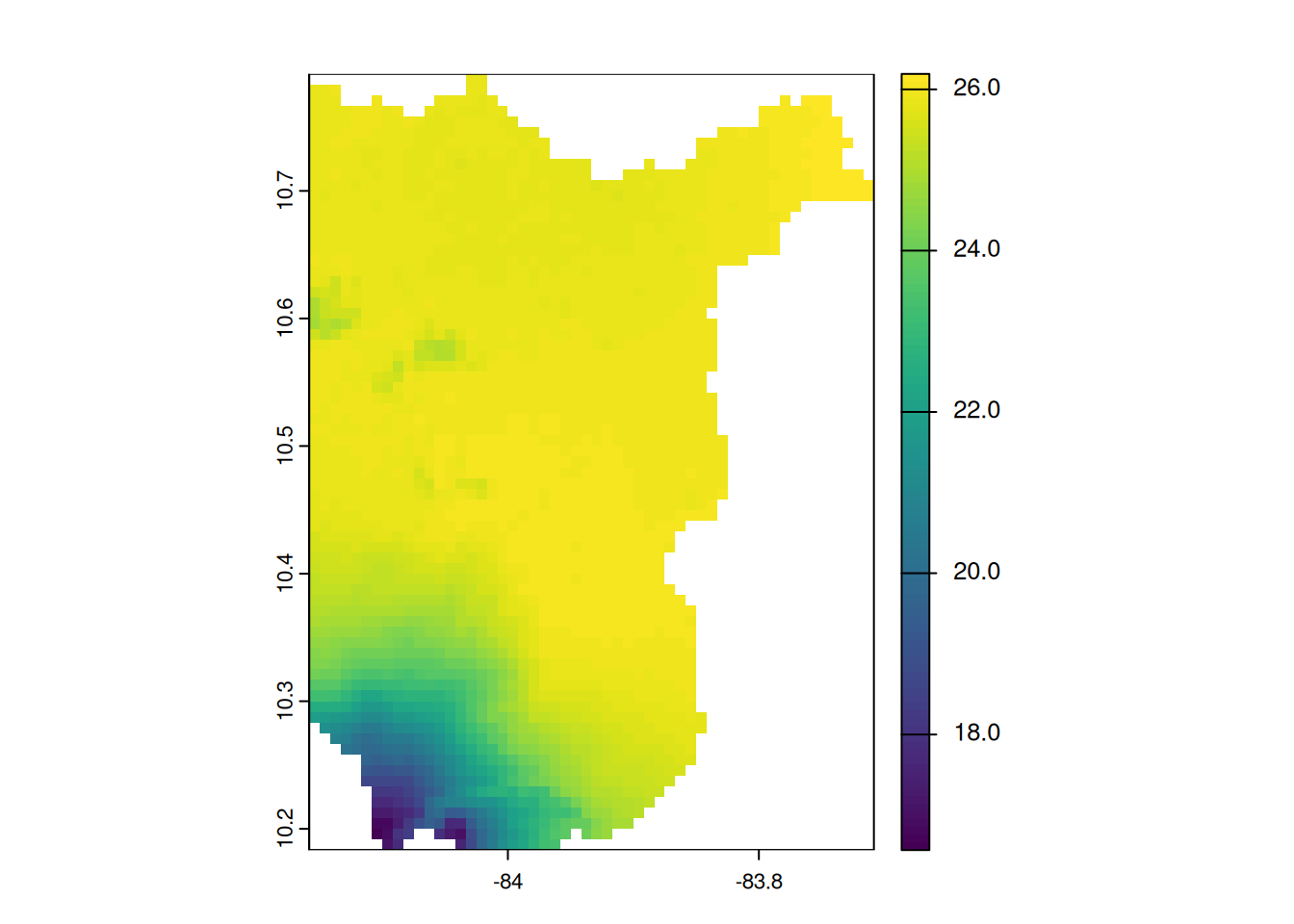
La función mask() “enmascara” (asigna NA) las celdas que el polígono no cubre. Es decir, respeta exactamente la forma del polígono y “borra” todo lo demás.
13.7.1.2 Álgebra de mapas
El álgebra de mapas permite crear nuevas capas espaciales al combinar y transformar capas existentes mediante operaciones matemáticas y lógicas aplicadas celda por celda, como es muestra en la Figura 13.2.
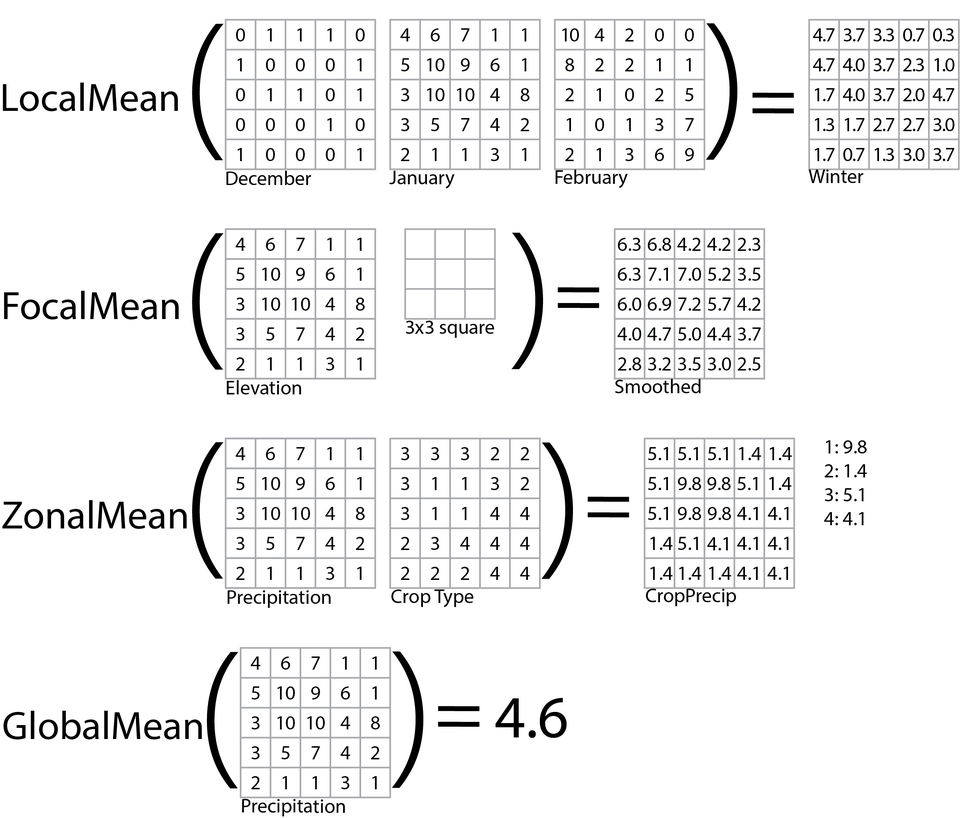
Cada capa se trata como una matriz en la que cada celda (o píxel) representa una propiedad geográfica específica (ej. elevación, temperatura, uso del suelo), de modo que operaciones simples (suma, resta, multiplicación, división) o funciones más complejas (estadísticas focales, reescalamientos, condicionales y funciones de vecindad) se ejecutan de forma sistemática sobre conjuntos de celdas análogas. El álgebra de mapas raster permite modelar procesos ambientales, evaluar idoneidad territorial, analizar riesgos y realizar simulaciones espaciales de manera reproducible y eficiente.
13.7.1.2.1 Ejemplo: cálculo del NDVI
El Índice de vegetación de diferencia normalizada (NDVI, en inglés, Normalized Difference Vegetation Index) es un indicador espectral que cuantifica la densidad y el vigor de la vegetación. Puede calcularse a partir de datos recopilados mediante teledetección.
13.7.1.2.1.1 Teledetección
La teledetección es la disciplina que trata sobre la adquisición de información sobre la superficie terrestre sin contacto físico directo, registrando la energía electromagnética (EM) reflejada o emitida por los objetos. Las formas más comunes de teledetección se basan en energía electromagnética reflejada. Cuando la energía (luz) del sol u otra fuente golpea un objeto, se refleja una parte de la energía. Diferentes materiales reflejan diferentes cantidades de energía entrante, lo que permite identificar los objetos. Por ejemplo, la energía que refleja una zona boscosa es diferente a la que refleja un cultivo o una ciudad, como se ilustra en la Figura 13.3.
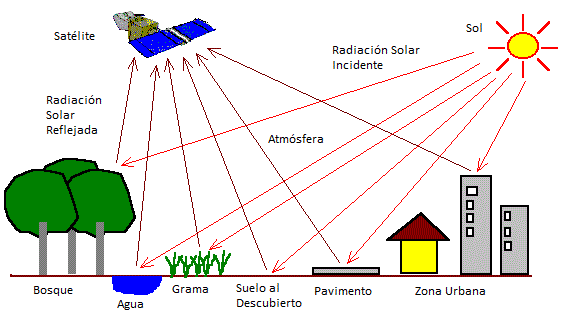
Los sensores instalados en satélites (y aviones o drones) “observan” distintas porciones del espectro electromagnético, mediante bandas discretas. Cada banda registra la intensidad de la señal en un rango de longitudes de onda y, por tanto, resalta propiedades físicas particulares. Así, los satélites orbitan la Tierra con cámaras multiespectrales (ej. Landsat 8/9 OLI, Sentinel-2 MSI) o hiperespectrales (ej. EnMAP, PRISMA) que dividen el espectro en decenas o cientos de bandas, además de sensores térmicos (ej. Landsat TIRS, MODIS) y de radar de apertura sintética (SAR) activos en microondas (ej. Sentinel-1, SAOCOM). Cada banda cumple un papel específico: distinguir vegetación sana (rojo + NIR), identificar humedad del suelo (SWIR), estimar temperatura (térmico), penetrar nubes y medir estructura (SAR), etc. La combinación de estas bandas mediante álgebra de mapas raster, índices espectrales o clasificación multibanda permite cartografiar uso del suelo, monitorear cultivos, evaluar desastres y estudiar procesos planetarios con un nivel de detalle y cobertura imposible de lograr in situ.
Por ejemplo, los satélites Sentinel-2 (Sentinel-2A y Sentinel-2B) de la Agencia Espacial Europea constituyen una constelación multiespectral equipada con el instrumento MultiSpectral Instrument (MSI), capaz de registrar 13 bandas distribuidas en el espectro visible, el infrarrojo cercano (NIR) y el infrarrojo de onda corta (SWIR), con resoluciones espaciales de 10 m, 20 m y 60 m según la banda, lo que les permite monitorizar con alta frecuencia y detalle procesos terrestres tan diversos como la salud de la vegetación, la humedad del suelo, la presencia de aerosoles o las nubes cirros.
Las siguientes son las bandas de los satélites Sentinel-2:
| Banda | Longitud de onda central (nm) | Ancho de banda (nm) | Resolución (m) | Uso principal |
|---|---|---|---|---|
| B01 (aerosoles) | 443 | 20 | 60 | Detección de aerosoles, calibración atmosférica |
| B02 (azul) | 490 | 65 | 10 | Diferenciación suelo–vegetación, penetración agua clara |
| B03 (verde) | 560 | 35 | 10 | Contraste agua limpia/turbia, realce de vegetación |
| B04 (rojo) | 665 | 30 | 10 | Índices de vegetación (con NIR), usos/coberturas |
| B05 (borde rojo 1) | 705 | 15 | 20 | Fluorescencia clorofílica, estrés vegetal |
| B06 (borde rojo 2) | 740 | 15 | 20 | Clasificación de vegetación, contenido de clorofila |
| B07 (borde rojo 3) | 783 | 20 | 20 | Biomasa, estructura foliar |
| B08 (NIR) | 842 | 115 | 10 | NDVI/EVI, biomasa, delineación de costas |
| B8A (NIR estrecho) | 865 | 20 | 20 | Fotofisiología, discriminación de tipos de cultivo |
| B09 (vapor de agua) | 945 | 20 | 60 | Contenido atmosférico de H2O |
| B10 (cirros) | 1375 | 30 | 60 | Detección de nubes cirros |
| B11 (SWIR 1) | 1610 | 90 | 20 | Humedad de vegetación y suelo, separación nieve/nube |
| B12 (SWIR 2) | 2190 | 180 | 20 | Incendios, mineralogía, humedad del suelo |
13.7.1.2.1.2 NDVI
El NDVI mide el estado de la vegetación mediante la combinación matemática de la reflectancia registrada en las bandas rojo (R) e infrarrojo cercano (NIR) de una imagen satelital o aérea, de acuerdo con la fórmula NDVI = (NIR − R) / (NIR + R).
\[ \text{NDVI} \;=\; \frac{\text{NIR} - \text{R}}{\text{NIR} + \text{R}} \]
Los pigmentos clorofílicos absorben gran parte de la luz roja para la fotosíntesis, mientras que la estructura interna de las hojas refleja intensamente el NIR; por ello, valores altos de NDVI (cercanos a +1) se asocian con vegetación densa y saludable, valores cercanos a 0 indican cobertura escasa o suelos expuestos, y valores negativos reflejan superficies como agua, nieve o nubes. Esta métrica, ampliamente utilizada en teledetección y monitoreo ambiental, permite evaluar la productividad de cultivos, detectar estrés hídrico, seguir la fenología de ecosistemas y analizar cambios en la cobertura vegetal a diversas escalas temporales y espaciales, constituyéndose en una herramienta clave para la gestión de recursos naturales y la investigación climática.
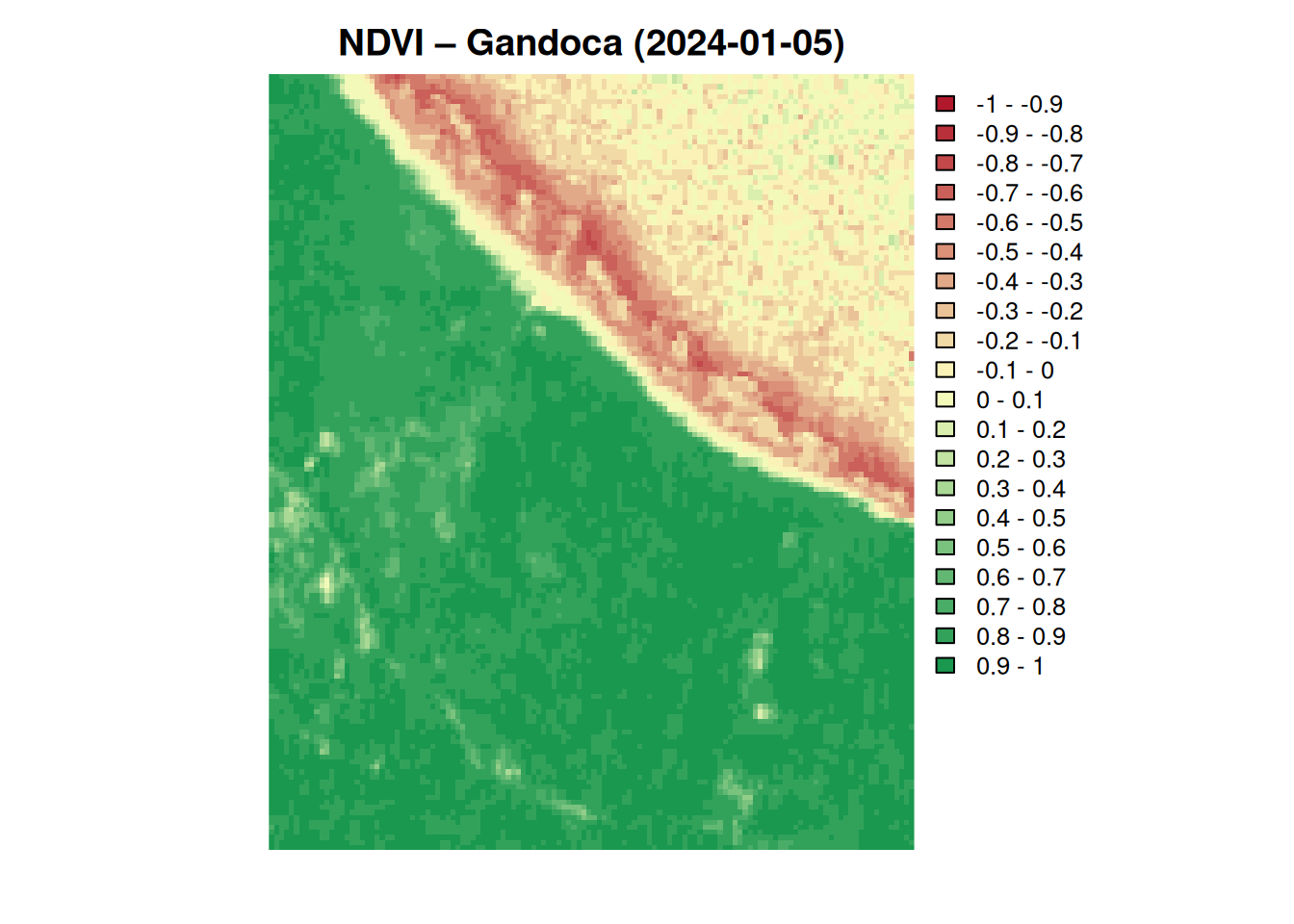
Seguidamente, se calcula el NDVI para una imagen Sentinel-2, tomada en Gandoca-Manzanillo el 2024-01-05.
Primero, se carga la imagen y se despliega en “color real” (true color).
Código
# Cargar imagen
imagen_gandoca_t1 = rast(
"https://github.com/gf0604-procesamientodatosgeograficos/2025-i/raw/refs/heads/main/datos/sentinel/gandoca-20240105.tif"
)
# Desplegar la imagen en "color real": bandas 4 (R), 3 (G) y 2 (B)
plotRGB(
imagen_gandoca_t1,
r = 4, # banda roja
g = 3, # banda verde
b = 2, # banda azul
stretch = "lin", # "lin" = estiramiento lineal; "hist" proporciona más contraste
main = "NDVI – Gandoca (2024-01-05)"
)
Luego se calcula el NDVI y se despliega el resultado.
Código
# Función para calcular el NDVI (red = banda roja, nir = banda infraroja cercana)
calcular_ndvi <- function(nir, red) {
(nir - red) / (nir + red)
}
ndvi_pal <- function(n = 100) {
colorRampPalette(
c("#b2182b", # rojo intenso (-1)
"#ffffbf", # amarillo ( 0)
"#1a9850") # verde profundo (+1)
)(n)
}
# Aplicar calcular_ndvi() sobre todas las celdas de la capa raster
imagen_gandoca_t1_ndvi <- lapp(
imagen_gandoca_t1[[c(8, 4)]],
fun = calcular_ndvi
)
# Desplegar NDVI
plot(
imagen_gandoca_t1_ndvi,
col = ndvi_pal(20), # 20 colores
breaks = seq(-1, 1, length.out = 21), # 20 clases iguales
zlim = c(-1, 1), # escala fija
legend = TRUE,
axes = FALSE, # como plotRGB()
box = FALSE,
main = "NDVI – Gandoca (2024-01-05)"
)
Ambas capas se muestran en el siguiente mapa interactivo:
Código
# Biblioteca para mapas interactivos
library(leaflet)
# Paleta continua de 10 colores
ndvi_colors <- colorRampPalette(c("#b2182b", "#ffffbf", "#1a9850"))(10)
pal_ndvi <- colorNumeric(
palette = ndvi_colors,
domain = c(-1, 1),
na.color = "transparent"
)
# Mapa leaflet
leaflet(options = leafletOptions(minZoom = 5)) |>
# Capas base
addProviderTiles("OpenStreetMap", group = "OpenStreetMap") |>
addProviderTiles("Esri.WorldImagery", group = "Esri World Imagery") |>
# Capa NDVI
addRasterImage(
imagen_gandoca_t1_ndvi,
colors = pal_ndvi,
opacity = 0.8,
project = FALSE,
group = "NDVI"
) |>
## Leyenda
addLegend(
pal = pal_ndvi,
values = c(-1, 1),
title = "NDVI",
position = "bottomright",
labFormat = labelFormat(digits = 1),
opacity = 1
) |>
## Escala métrica
addScaleBar(
position = "bottomleft",
options = scaleBarOptions(imperial = FALSE)
) |>
## Control de capas
addLayersControl(
baseGroups = c("OpenStreetMap", "Esri World Imagery"),
overlayGroups = c("NDVI"),
options = layersControlOptions(collapsed = FALSE)
)Ejercicio:
Genere una capa con el cálculo del NDVI para la imagen de Gandoca-Manzanillo tomada el 2024-06-18. Inclúyala en un mapa interactivo que permita comparar el cambio en la vegetación desde el 2024-01-05.