10 Tidyverse: colección de paquetes para ciencia de datos
10.1 Trabajo previo
10.1.1 Lecturas
Wickham, H., & Grolemund, G. (2017). R for Data Science: Import, Tidy, Transform, Visualize, and Model Data. O’Reilly Media. https://r4ds.had.co.nz/
10.2 Resumen
En este capítulo se estudia Tidyverse, una colección de paquetes para ciencia de datos.
10.3 Características generales
Tidyverse es una colección de paquetes de R enfocados en ciencia de datos. La ciencia de datos es una discipina que permite convertir datos no procesados en entendimiento, comprensión y conocimiento.4
10.3.1 Modelo de ciencia de datos
La figura 10.1 ilustra el modelo de un proyecto típico de ciencia de datos, el cual incluye los procesos de importar, organizar, transformar, visualizar, modelar y comunicar.
.](img/modelo-ciencia-datos.png)
Figure 10.1: Modelo de ciencia de datos. Imagen de Hadley Wickham.
Importar los datos, típicamente implica leerlos de un archivo, una base de datos o una interfaz de programación de aplicaciones (API) y cargarlos en un data frame. Organizar (to tidy) los datos significa colocarlos en estructuras rectangulares, similares a tablas, de manera que cada fila sea una observación y cada columna una variable.
Una vez que los datos han sido importados y organizados, suele ser necesario realizar en estos algún tipo de transformación. Transformar los datos puede implicar la generación de algún subconjunto de filas y columnas, la creación de nuevas variables o el cálculo de estadísticas (ej. conteos, promedios).
Cuando los datos han sido organizados con las variables necesarias, pueden aplicarse dos fuentes de generación de conocimiento: la visualización y el modelado. Al visualizar los datos (en tablas, gráficos, mapas, etc.), pueden encontrarse patrones inesperados o pueden surgir nuevas preguntas. Por su parte, los modelos son herramientas matemáticas o computacionales que facilitan la descripción o predicción de un problema.
El último paso es la comunicación, una actividad crítica de cualquier proyecto de análisis de datos o de ciencia en general. Todos estos procesos se articulan mediante programación de computadoras.
Los paquetes de Tidyverse “comparten filosofía de diseño, gramática y estructuras de datos”5 para apoyar estos procesos del modelo de ciencia de datos. El concepto de Tidyverse fue introducido por Hadley Wickham, quien también ha programado varios de sus paquetes.
10.3.2 Paquetes
El núcleo de Tidyverse está compuesto por ocho paquetes base, los cuales proveen las funcionalidades utilizadas más frecuentemente en análisis de datos:
- ggplot2: sistema para la creación declarativa de gráficos, basado en el libro The Grammar of Graphics, de Wilkinson et al..
- dplyr: gramática para la manipulación de datos que proporciona un conjunto consistente de “verbos” que resulven los retos más comunes de manipulación de datos.
- tidyr: conjunto de funciones para organizar (to tidy) datos, colocando las observaciones, variables y valores en filas, columnas y celdas de estructuras rectangulares.
- readr: conjunto de funciones para cargar datos de estructuras rectangulares (ej. archivos CSV) en memoria.
- purr: conjunto de herramientas de programación funcional para trabajar con funciones y vectores.
- tibble: un tibble es una redefinición del concepto de data frame, para hacerlos más eficientes y fáciles de usar.
- stringr: colección de funciones para facilitar el trabajo con hileras de caracteres.
- forcats: colección de funciones para facilitar el trabajo con factores.
Hay otros paquetes para tareas más especifícas relacionadas con importación, limpieza y modelado de datos, entre otras. Los paquetes de Tidyverse son de los más descargados, entre la totalidad de paquetes del lenguaje de programación R.
10.4 Instalación y carga
Los paquetes de Tidyverse pueden instalarse de manera conjunta o individualmente con la función install.packages():
# Instalación conjunta
install.packages("tidyverse")
# Instalación de paquetes individuales
install.packages("ggplot2")
install.packages("dplyr")Una vez instalados, los paquetes también pueden cargarse conjunta o separadamente con la función library():
# Carga conjunta
library(tidyverse)
# Carga de paquetes individuales
library(ggplot2)
library(dplyr)También es posible utilizar la notación paquete::funcion() para llamar una función sin necesidad de cargar todo el paquete:
# Llamado a la función glimpse() del paquete dplyr
dplyr::glimpse(iris)
#> Rows: 150
#> Columns: 5
#> $ Sepal.Length <dbl> 5.1, 4.9, 4.7, 4.6, 5.0, 5.4, 4.6, 5.…
#> $ Sepal.Width <dbl> 3.5, 3.0, 3.2, 3.1, 3.6, 3.9, 3.4, 3.…
#> $ Petal.Length <dbl> 1.4, 1.4, 1.3, 1.5, 1.4, 1.7, 1.4, 1.…
#> $ Petal.Width <dbl> 0.2, 0.2, 0.2, 0.2, 0.2, 0.4, 0.3, 0.…
#> $ Species <fct> setosa, setosa, setosa, setosa, setos…
10.5 El conjunto de datos palmerpenguins
palmerpenguins es un paquete de datos de R ampliamente utilizado para ejemplificar funciones de exploración y visualización. Es muy popular en ciencia de datos en general y también está disponible para otros lenguajes de programación (ej. Python, Julia). Se utiliza como una alternativa a otros conjuntos de datos como, por ejemplo, iris.
Los datos de palmerpenguins fueron recolectados entre 2007 y 2009 por la Dr. Kristen Gorman y el Programa de Investigación Ecológica de Largo Plazo (LTER) de la Estación Palmer. Consisten de 344 observaciones de pingüinos de tres especies, las cuales habitan en tres islas del archipiélago Palmer, en la Antártida. Para cada individuo se registraron variables como especie, sexo, masa (peso), longitud de la aleta (flipper), longitud del pico (bill) y profundidad del pico, entre otras.
En R, el paquete puede instalarse con la función install.packages():
# Instalación de palmerpenguins
install.packages("palmerpenguins")Una vez instalado, el paquete puede cargarse con la función library():
El paquete contiene dos conjuntos de datos:
- penguins_raw: datos sin procesar.
- penguins: subconjunto curado de los datos sin procesar.
palmerpinguins se utilizará en este capítulo, y en los siguientes, para ejemplificar varias de las funcionalidades de Tidyverse.
10.5.1 Ejemplos de visualizaciones
Se muestran varios tipos de gráficos estadísticos generados con la función ‘ggplot()’.
10.5.1.1 Gráficos de dispersión
Este tipo de gráficos muestra relaciones entre variables.
# Gráfico de dispersión de longitud del pico vs masa (peso)
ggplot(data = penguins, aes(x = bill_length_mm, y = body_mass_g)) +
geom_point(size = 2) +
geom_smooth(method = "lm", se = FALSE) +
ggtitle("Longitud del pico vs. masa") +
xlab("Longitud del pico (mm)") +
ylab("Masa (g)") +
labs(color = "Especie", shape = "Especie")
#> `geom_smooth()` using formula 'y ~ x'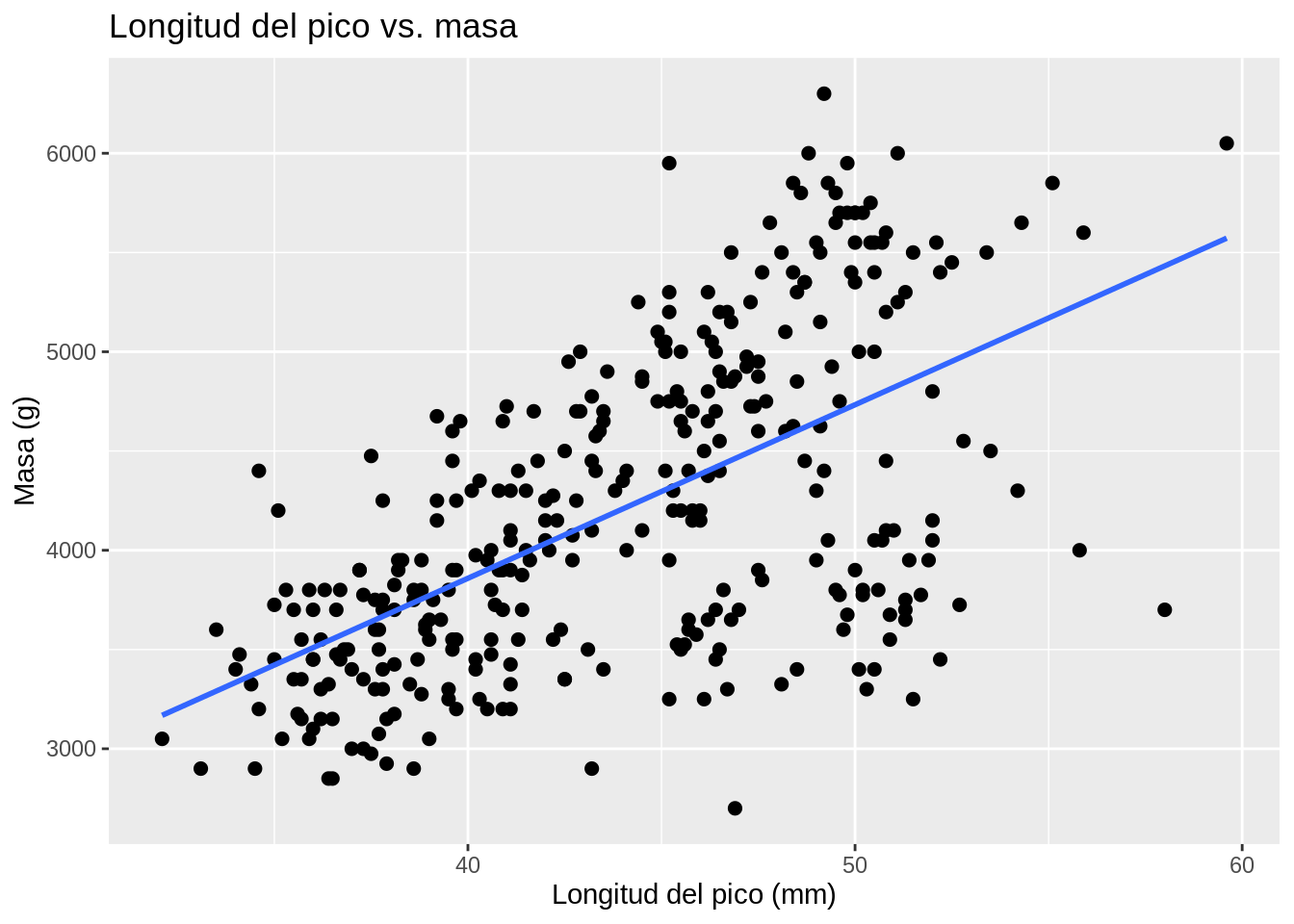
# Gráfico de dispersión de longitud del pico vs masa (peso) por especie
ggplot(data = penguins, aes(x = bill_length_mm, y = body_mass_g)) +
geom_point(aes(color = species,
shape = species),
size = 2) +
geom_smooth(method = "lm", se = FALSE, aes(color = species)) +
scale_color_manual(values = c("darkorange", "darkorchid", "cyan4")) +
ggtitle("Longitud del pico vs. masa por especie") +
xlab("Longitud del pico (mm)") +
ylab("Masa (g)") +
labs(color = "Especie", shape = "Especie")
#> `geom_smooth()` using formula 'y ~ x'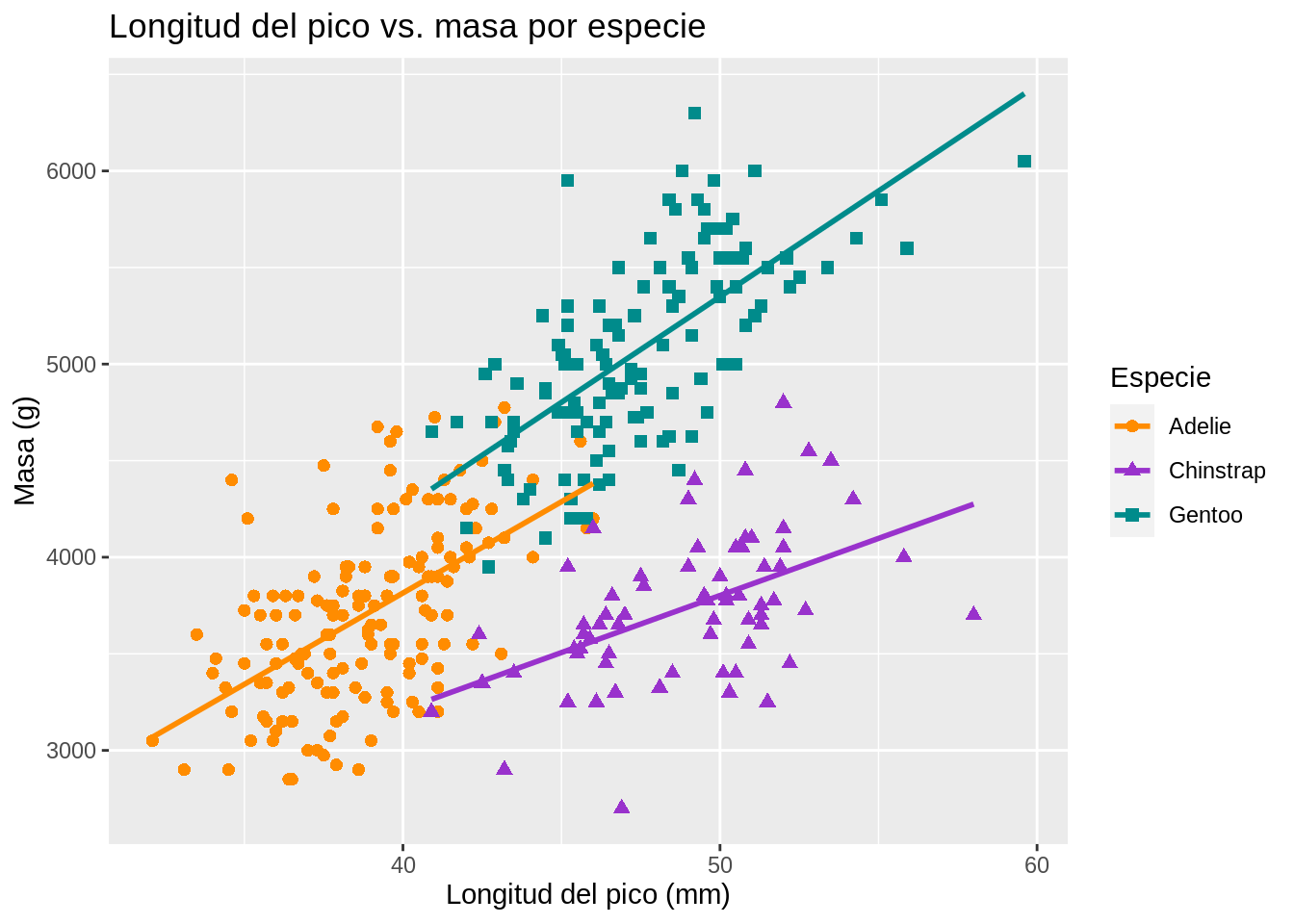
10.5.1.2 Histogramas
Este tipo de gráficos muestra distribuciones de variables.
# Distribución de la variable de masa (peso)
ggplot(data = penguins, aes(x = body_mass_g)) +
geom_histogram() +
ggtitle("Distribución de la variable masa (peso)") +
xlab("Masa (g)") +
ylab("n")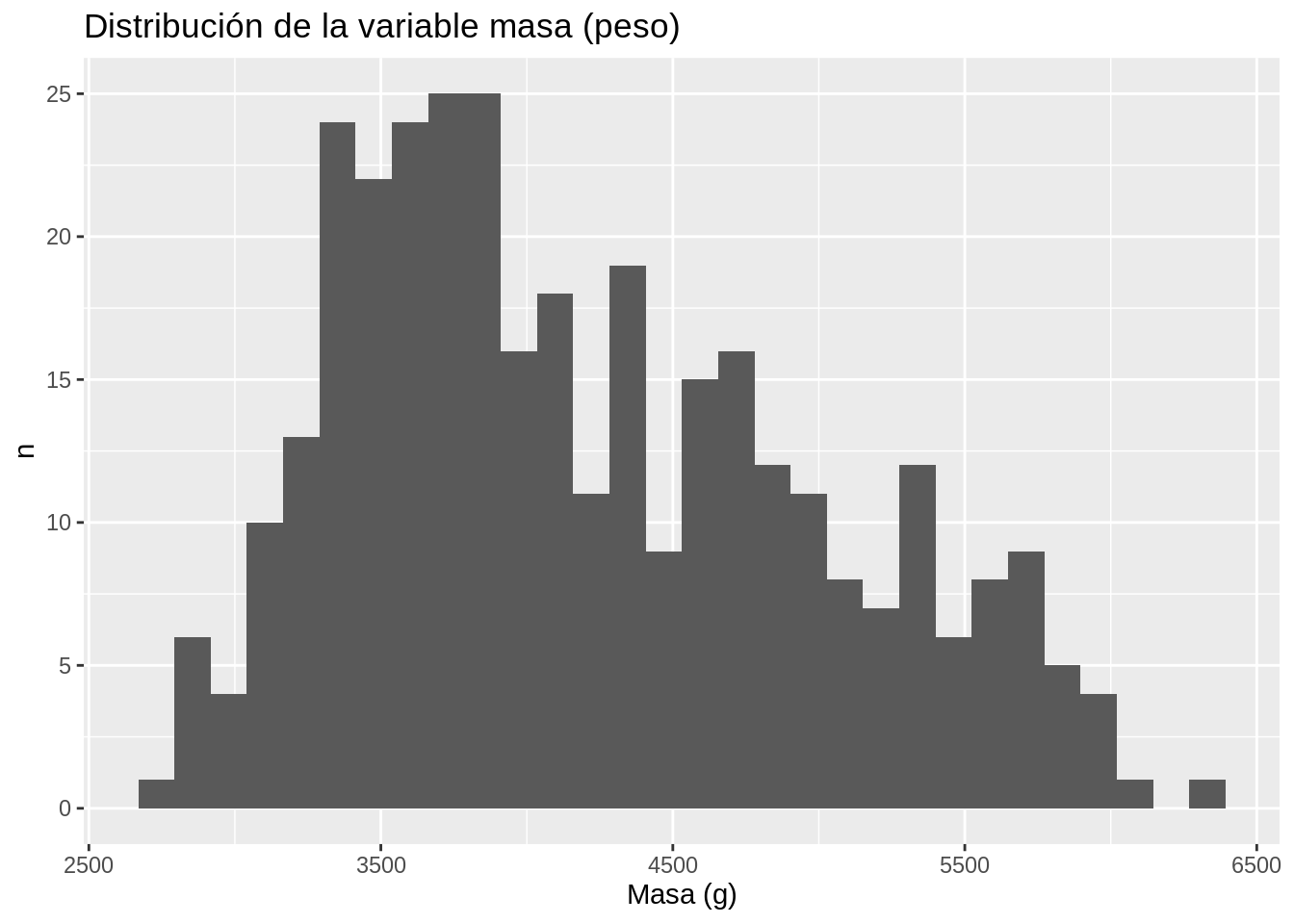
# Distribución de la variable de masa (peso) por especie
ggplot(data = penguins, aes(x = body_mass_g)) +
geom_histogram(aes(fill = species), alpha = 0.5, position = "identity") +
scale_fill_manual(values = c("darkorange","darkorchid","cyan4")) +
ggtitle("Distribución de la variable masa (peso) por especie") +
xlab("Masa (g)") +
ylab("n") +
labs(fill = "Especie")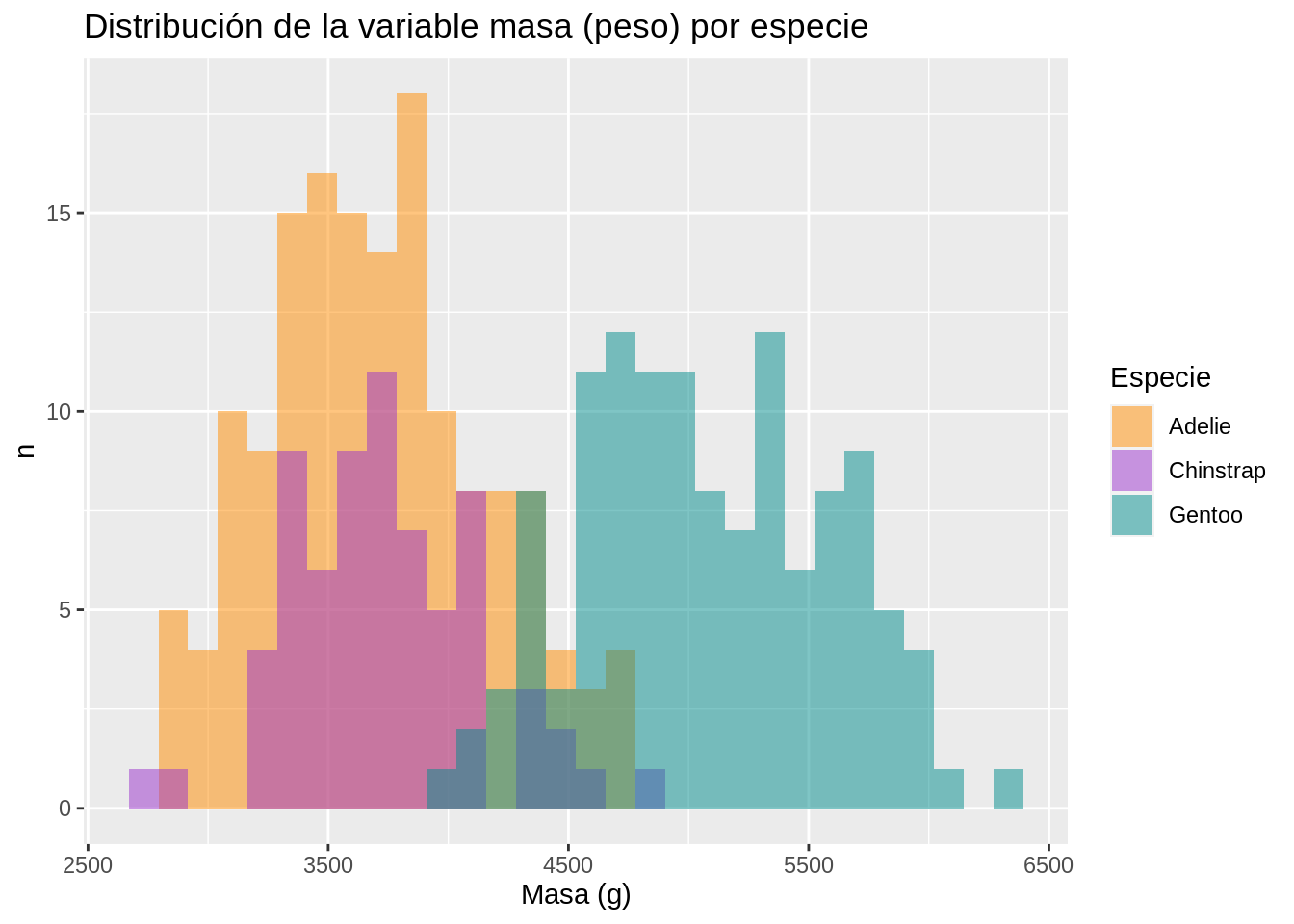
10.5.1.3 Diagramas de caja
Este tipo de gráficos muestra datos a través de sus cuartiles.
# Diagrama de caja de la variable masa (peso)
ggplot(data = penguins, aes(y = body_mass_g)) +
geom_boxplot() +
ylab("Masa (g)")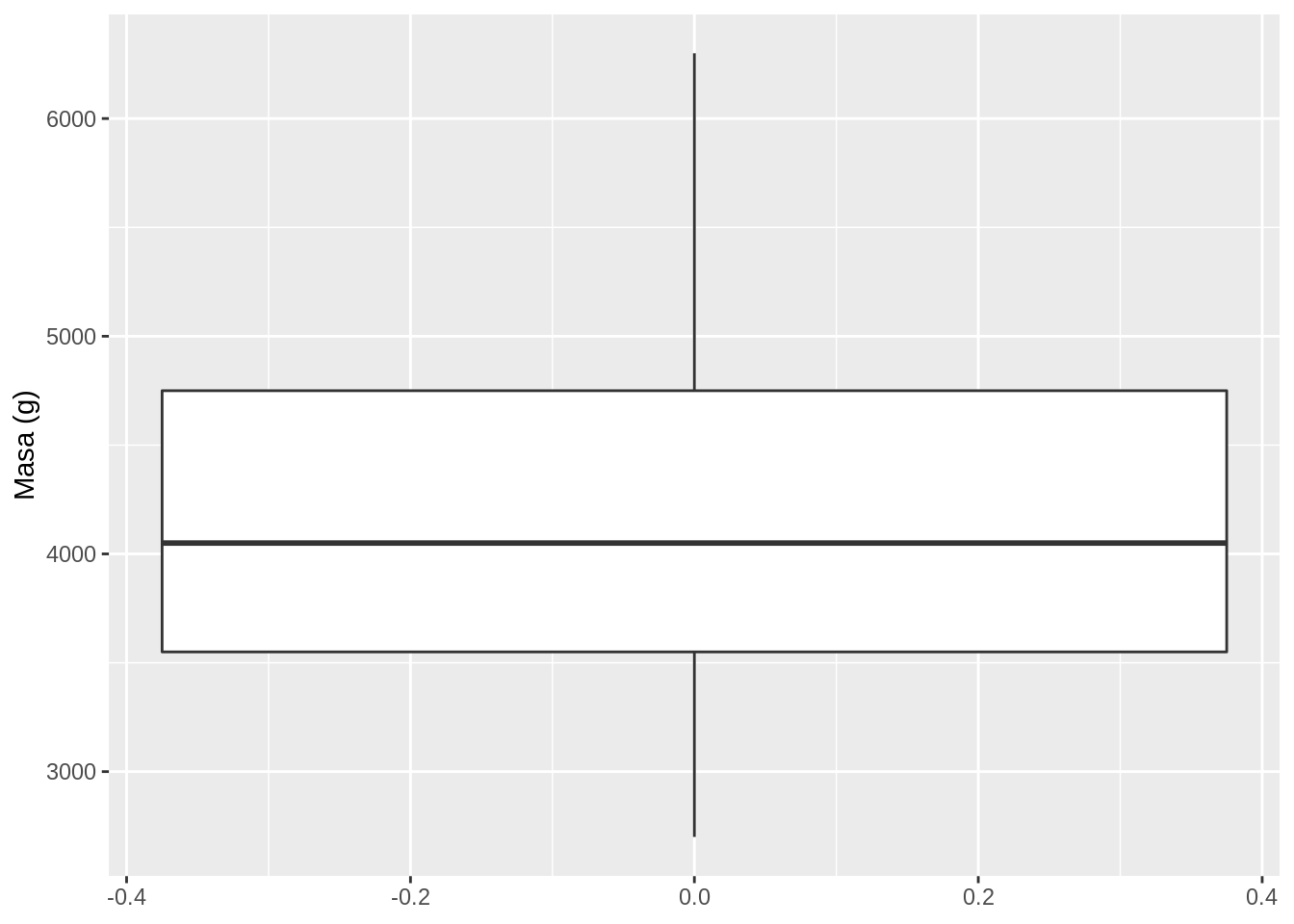
# Diagrama de caja de la variable masa (peso) por especie
ggplot(data = penguins, aes(x = species, y = body_mass_g)) +
geom_boxplot(aes(color = species), width = 0.3, show.legend = FALSE) +
scale_color_manual(values = c("darkorange","purple","cyan4")) +
xlab("Especie") +
ylab("Masa (g)")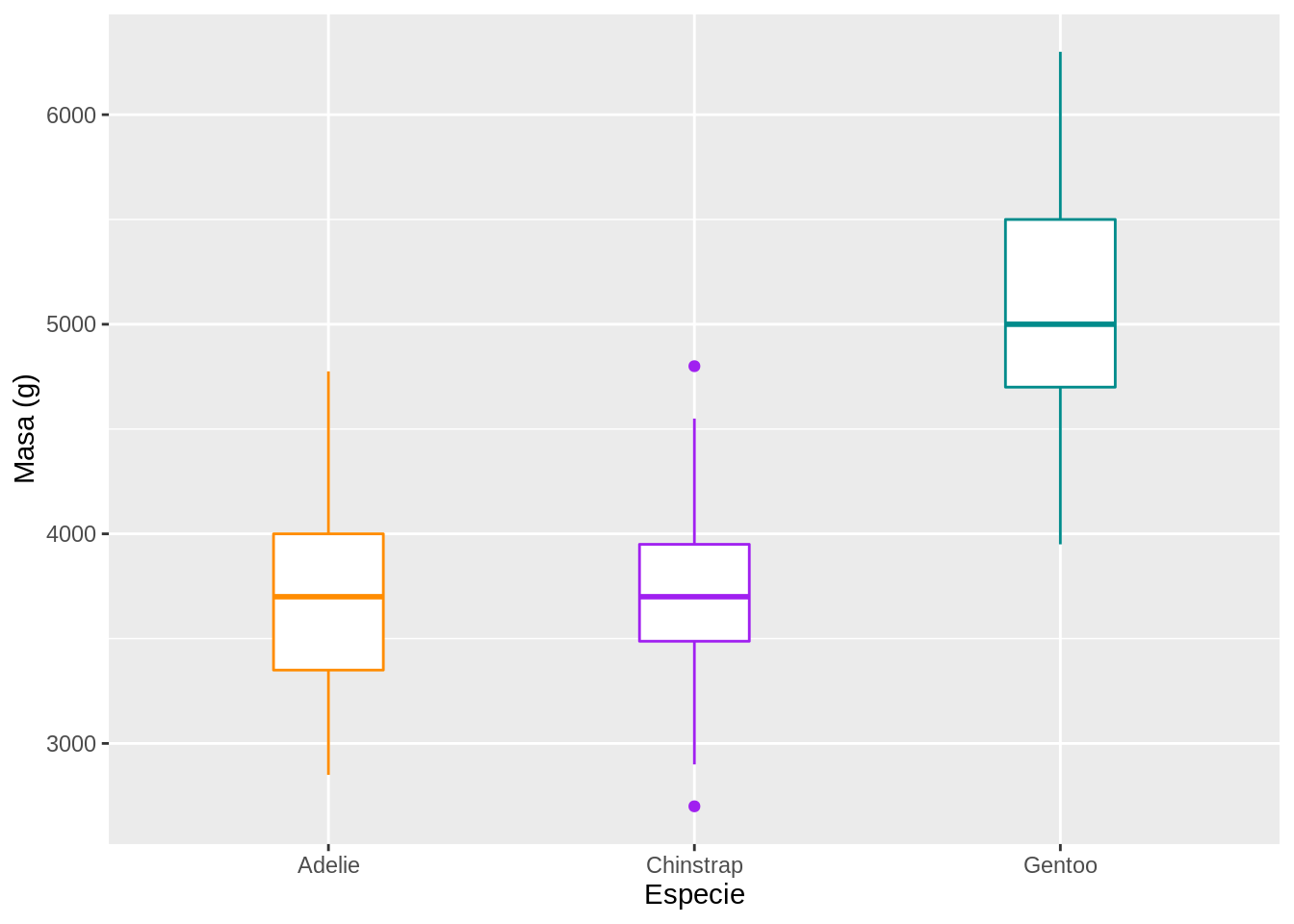
10.6 Datos tidy
Los paquetes de Tidyverse trabajan con datos tidy (i.e. ordenados, organizados), un concepto también introducido por Hadley Wickham y que está relacionado con la organización de los datos en estructuras rectangulares de filas y columnas, similares a las tablas o matrices.
Según Wickham, los datos tidy deben cumplir con tres características:
- Cada variable debe tener su propia columna.
- Cada observación debe tener su propia fila.
- Cada valor debe tener su propia celda.
Estas características se ilustran en la figura 10.2.
.](img/datos-tidy.png)
Figure 10.2: Datos tidy. Imagen de Hadley Wickham.
El empleo de este modelo de datos es común en todos los paquetes de Tidyverse, lo que posibilita aprender y usar sus funciones con mayor facilidad. Además, permite invertir menos esfuerzo en lidiar con diferentes modelos de datos y así dedicar más tiempo y esfuerzo en los problemas de análisis a resolver.
10.6.0.1 Tibbles
Los datos tidy pueden almacenarse en los tradicionales data frames (tipo data.frame) de R. Adicionalmente, Tidyverse implementa el tipo de datos tibble o tbl. Un tbl también es un data.frame, pero más “liviano” y fácil de usar.
Una de las diferencias entre un data.frame y un tibble es el despliegue de los datos:
# Clase del conjunto de datos penguins
class(penguins)
#> [1] "tbl_df" "tbl" "data.frame"
# Impresión del tibble penguins
print(penguins)
#> # A tibble: 344 × 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Adelie Torgersen 39.1 18.7
#> 2 Adelie Torgersen 39.5 17.4
#> 3 Adelie Torgersen 40.3 18
#> 4 Adelie Torgersen NA NA
#> 5 Adelie Torgersen 36.7 19.3
#> 6 Adelie Torgersen 39.3 20.6
#> 7 Adelie Torgersen 38.9 17.8
#> 8 Adelie Torgersen 39.2 19.6
#> 9 Adelie Torgersen 34.1 18.1
#> 10 Adelie Torgersen 42 20.2
#> # … with 334 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>Como puede verse, la función print() despliega solamente las 10 primeras filas del tibble y una cantidad limitada de columnas, mostrando así una salida más legible. Este comportamiento puede modificarse con los argumentos de print().
# Impresión del tibble penguins con 15 filas y todas las columnas
print(penguins, n=15, width = Inf)
#> # A tibble: 344 × 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Adelie Torgersen 39.1 18.7
#> 2 Adelie Torgersen 39.5 17.4
#> 3 Adelie Torgersen 40.3 18
#> 4 Adelie Torgersen NA NA
#> 5 Adelie Torgersen 36.7 19.3
#> 6 Adelie Torgersen 39.3 20.6
#> 7 Adelie Torgersen 38.9 17.8
#> 8 Adelie Torgersen 39.2 19.6
#> 9 Adelie Torgersen 34.1 18.1
#> 10 Adelie Torgersen 42 20.2
#> 11 Adelie Torgersen 37.8 17.1
#> 12 Adelie Torgersen 37.8 17.3
#> 13 Adelie Torgersen 41.1 17.6
#> 14 Adelie Torgersen 38.6 21.2
#> 15 Adelie Torgersen 34.6 21.1
#> flipper_length_mm body_mass_g sex year
#> <int> <int> <fct> <int>
#> 1 181 3750 male 2007
#> 2 186 3800 female 2007
#> 3 195 3250 female 2007
#> 4 NA NA <NA> 2007
#> 5 193 3450 female 2007
#> 6 190 3650 male 2007
#> 7 181 3625 female 2007
#> 8 195 4675 male 2007
#> 9 193 3475 <NA> 2007
#> 10 190 4250 <NA> 2007
#> 11 186 3300 <NA> 2007
#> 12 180 3700 <NA> 2007
#> 13 182 3200 female 2007
#> 14 191 3800 male 2007
#> 15 198 4400 male 2007
#> # … with 329 more rows10.7 Pipes
Las funciones de Tidyverse pueden encadenarse a través del operador pipe (%>%) (tubo), para formar pipelines (tuberías). En este contexto, un pipeline consiste de una cadena de procesos conectados de forma tal que la salida de cada proceso de la cadena es la entrada del próximo. Esto permite la comunicación y sincronización entre los procesos y evita la anidación (nesting) de llamados a funciones.
El siguiente ejemplo implementa un pipeline de funciones de Tidyverse:
# Cadena de "pipes" entre funciones de Tidyverse
penguins %>%
dplyr::filter(species == "Gentoo") %>% # subconjunto de observaciones
select(species, bill_length_mm, flipper_length_mm) # subconjunto de columnas
#> # A tibble: 124 × 3
#> species bill_length_mm flipper_length_mm
#> <fct> <dbl> <int>
#> 1 Gentoo 46.1 211
#> 2 Gentoo 50 230
#> 3 Gentoo 48.7 210
#> 4 Gentoo 50 218
#> 5 Gentoo 47.6 215
#> 6 Gentoo 46.5 210
#> 7 Gentoo 45.4 211
#> 8 Gentoo 46.7 219
#> 9 Gentoo 43.3 209
#> 10 Gentoo 46.8 215
#> # … with 114 more rowsUna alternativa a los pipes es la anidación de llamados a funciones:
# Llamados anidados a funciones
select(filter(penguins, species == "Gentoo"),
bill_length_mm,
flipper_length_mm)
#> # A tibble: 124 × 2
#> bill_length_mm flipper_length_mm
#> <dbl> <int>
#> 1 46.1 211
#> 2 50 230
#> 3 48.7 210
#> 4 50 218
#> 5 47.6 215
#> 6 46.5 210
#> 7 45.4 211
#> 8 46.7 219
#> 9 43.3 209
#> 10 46.8 215
#> # … with 114 more rowsEl uso de pipes permite un funcionamiento homogéneo de las funciones de Tidyverse:
- El primer argumento es un data frame. Puede omitirse si la función recibe el data frame a través del operador pipe.
- Los argumentos siguientes describen que hacer con el data frame, utilizando los nombres de las columnas (sin comillas).
- El resultado es un nuevo data frame.
10.8 Recursos de interés
Canelón, S. (2020). An Antarctic Tour of the Tidyverse. https://spcanelon.github.io/tour-of-the-tidyverse/
R- Ladies Global. (2020). R-Ladies Chicago (English)—An Antarctic Tour of the Tidyverse—Silvia Canelón. https://www.youtube.com/watch?v=m_ZoMmAIx-o
Wickham, H. (2014). Tidy Data. Journal of Statistical Software, 59(1), 1-23. https://doi.org/10.18637/jss.v059.i10
Wickham, H., & Grolemund, G. (s. f.). R para Ciencia de Datos. Recuperado 14 de mayo de 2022, de https://es.r4ds.hadley.nz/